土壌細菌の遺伝生態研究の現状
-ダイズ根粒菌の実例をまじえて-
南澤 究
東北大学大学院生命科学研究科
〒980-8577 宮城県仙台市青葉区片平2_1_1
Progresses and Perspectives of Genetic Ecology of Soil Microbes
Kiwamu Minamisawa
Institute of Genetic Ecology,
Tohoku University, Katahira, Aoba-ku, Sendai 980-8577, Japan
Key Words: Soil microbes, Bradyrhizobium,
Insertion sequence, Genome rearrangement,
Starvation
土壌細菌は地球レベルの物質循環や植物生育の上で大変重要な役割を果たしており、地球生態系の恒常性の維持に貢献している。また、環境食料問題の関連でも、組換え生物の利用の面でも土壌微生物は注目されている。しかし、土壌中に生息している細菌については、分からないことだらけである。例えば、土壌中に生息している細菌の中で培養可能なものは一部であり、生きているが培養困難である細菌細胞や数回分裂すると増殖が停止してしまう細胞が圧倒的に多い。土壌から分離された細菌の形質が、元の環境で機能していることが解明されている例は少ない。水田土壌や畑土壌において栄養共生や酸化還元を介した物質循環はどのような細菌コンソーシアムにより担われているのであろうか。
学問の歴史はくり返しており、微生物学や生物学は近代科学の名のものに一旦バラバラに分解されてきたが、今あらためて統合され自然界の微生物ダイナミズムをさらに深く理解する段階に近付いているのではないだろうか。このような統合の流れは、生物学全体で進んでおり、環境食糧問題と接点をもつ土壌微生物学の方からも、基本的な課題の見直しと、新たな研究展開の方向性を吟味すべき時期になっていると思われる。本稿では、著者が関わってきたダイズ根粒菌の遺伝生態研究を紹介しながら、土壌細菌の生活について考えてみたい。
2.Bradyrhizobium属ダイズ根粒菌の遺伝生態
1) 土壌細菌と共生窒素固定細菌としての生活環をもつ根粒菌
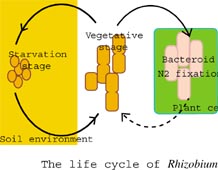
根粒菌は、マメ科植物に根粒を形成し、共生窒素固定を行う一群のグラム陰性の土壌細菌である。共生状態の根粒菌、すなわち根粒バクテロイドは、分子状窒素をアンモニアに変換する窒素固定作用を営み、農業生産や地球レベルの窒素循環に重要な役割を果 たしている。宿主植物がない場合、土壌中で単生生活をしているが、共生状態になると細胞内に多量 のニトロゲナーゼタンパクを蓄積し、共生窒素固定に適したエネルギー生産系・呼吸系・輸送系をもったバクテロイド細胞に分化する。根粒菌と宿主植物の間で、様々なシグナル交換があって初めて窒素固定活性のある根粒が完成する。老化した根粒は崩壊し、バクテロイドは再び単生生活の根粒菌となり土壌へ戻る。このように、根粒菌は土壌中の単生生活と宿主植物内における共生生活という二つの生活環をもった細菌である(図1)。特に、生育の遅いBradyrhizobium属根粒菌は、蒸留水中で1年以上も生存し1、2)、alpha-Proteobacteriaの低栄養細菌のグループと近縁であり3、4)、従属栄養条件のみでなく一部の株は化学独立栄養的に生育も可能であり、嫌気的条件下において異化的な硝酸還元(脱窒)能をもつなど、いわゆる土壌固有型細菌としての性質を有している3)。ダイズの起源地とされている中国、日本の農耕地土壌にはBradyrhizobium 属ダイズ根粒菌は普遍的に生息しているおり、土壌細菌の遺伝的多様性・地理的な分布、進化・環境適応を調べる上で、面 白い研究対象である。
図1根粒菌の生活環
2) Bradyrhizobium属ダイズ根粒菌の遺伝的多様性と風土性
ダイズ生産との関係では、優良根粒菌の接種が効かない「土着ダイズ根粒菌との競合問題」という背景があり、私どもは土着ダイズ根粒菌の遺伝的多様性を挿入配列(IS)によるRSフィンガープリント法により検討してきた。その結果 、一枚の圃場においても、極めて多様なフィンガープリントが観察された5)。RSフィンガープリントは、種やサブグループ毎に異なり、土着ダイズ根粒菌の進化を反映していた5)。さらに日本国内の6圃場を比較したところ、RSフィンガープリントの多様性やそのパターンには圃場間で偏りが存在し、いわゆる風土性(Endemism)が観察された(図2)。このような遺伝的多様性や風土性がどのように生成したか今のところ不明であるが、土壌条件や圃場管理の関与が考えられると同時に環境中における土壌細菌の進化などと関わりが考えられる3,6)。
図2 日本国内の5圃場(A-E)に生息しているダイズ根粒菌RSα、RSβ-フィンガープリント 8)
多数の挿入配列(IS)を保有したダイズ根粒菌HRS株が新潟の長倉圃場において多数見い出された(図2、Site A)。このHRS株は、通常のnon-HRS株と比較して培地(HM,YM)中における生育が極端に遅く(世代時間10時間以上)、その他の性質は、non-HRS株とほぼ同じであった。特に、根粒形成や窒素固定などの共生に関わる表現型は同様であった7)。しかし、培地を10倍、100倍と希釈して行くと、全体として生育速度は低下するが、両者の生育速度の違いも見られなくなった。したがって、土壌条件に近い低栄養環境下ではHRS株とnon-HRS株の生育は同程度であると考えられた。新潟の長倉圃場に栽培されているダイズ根粒の約半数は常にHRS株で形成されていた。この圃場は田畑輪環されているので、土壌をポットで湛水処理したところ、HRS株の出現頻度が有為に上昇した。したがって、土壌湛水条件下においてHRS株が生成している可能性が考えらえた。
新潟および十勝の国内の圃場のみでなく、米国のアルカリ土壌や中国から分離された生育の極端に遅いダイズ根粒菌もHRS株であることが最近分かってきた3, 8)。通常、Bradyrhizobium属ダイズ根粒菌の根粒形成遺伝子、窒素固定遺伝子などの共生領域は、ゲノム上に共生アイランドとして存在しているが、HRS株では共生領域の遺伝子間に多数のISが挿入され、かつ共生領域が分断され非共生領域も含めたシャフリングを起こしているようであった(未発表)。
HRS株とnon-HRS株の血清型および16SrRNA遺伝子配列が圃場毎に一致するので7)、HRS株はnon-HRS株からISの関与したゲノム再編成によって圃場生態系において生成していると考えられる(図3)。今のところ、ゲノム再編成の遺伝的な機構は不明であるが、このようなゲノムシャフリングは、HRS株ゲノムのみに存在するIS1631の感染と何らかの環境ストレスにより生成しているないかと考えている8)。HRS株は、通 常の高栄養の培地での生育が遅いので、今までの研究で見逃されてきたもので、土壌環境下で繰り広げられている細菌の進化および遺伝的多様化の一側面 を示しているのではないだろうか。最近、HRS株を高栄養条件下で培養した場合、細胞サイズが、non-HRS株より3割程度小さいことが分かりつつある。ダイズ根粒菌HRS株は、土壌細菌の多様化・飢餓応答・培養の困難性・ゲノム進化などの土壌微生物学の基本課題とつながる課題と材料を提供しているのではないかと考えている。そこで、次に、土壌環境中で細菌はどのような生活をしているかという点について考察してみたい。
図3 ダイズ根粒菌HRS株生成の仮説 7), 8)
3, 土壌中で細菌はどのような生活をしているか?
土壌に生息している細菌は実験室で培養されている細菌より小さく、その7割近くが0.3ミクロン以下の小型の細胞であると報告されている9)。共焦点蛍光顕微鏡で土壌細菌の細胞体積を正確に測定すると、その小型の細菌細胞が土壌バイオマスとほぼ一致する10)。外部から栄養基質を供給すると数日で細胞体積の増加や細胞分裂が観察されるので、これらの小型の細胞は生きており、物質循環等の機能を担っていると考えられる10)。根粒菌も土壌に定着する細菌であり、根粒形成能を保持した小型細胞が下層土で多いことが報告されている11)。栄養源が枯渇した定常期における大腸菌は、種々の環境ストレスに耐えうるように構造的・生理的に変化し、球菌状に小型化する12)。小型化した細胞は増殖期の細胞と比較して、外膜のリポ多糖の増加、細胞壁のペプチドクリカン層の増加、内膜のリン脂質の増加、核様体の凝縮、ペリプラズムへのトレハロースの蓄積などを起こし、大腸菌の場合、これらの変化はシグマ因子の関与するグローバルな転写制御が大きく関わっている12)。最近、水系においてもコレラ菌などの病原菌が「生きているが培養できない状態(VBNC)」になることが注目されており、自然環境下で、増殖せず、飢餓状態で生きている細菌の姿ではないかと考えられている13,14)。
このように土壌などの自然環境下に生息している細菌の増殖、飢餓応答、死滅は、土壌微生物学の基本課題の一つであると考えるが、その生理的、遺伝的な仕組みについてほとんど明らかになっていない。しかし、近年のゲノムレベルの研究や地道な生理学的なアプローチから突破口になりそうな面白い知見が報告されているので紹介したい。
近年、10種以上の細菌のゲノムの全塩基配列が明らかになり、生物のゲノムがどのように構成されているかという比較が可能となってきた。そこで浮かび上がってきた共通 の特徴として、複製と遺伝子の「向き」が同じになる傾向があることが分かってきた15,16)。ほとんどの細菌は、図4のような環状のゲノムであり、複製開始点から両側に複製フォークが進み、最終的にゲノム全体が複製され、隔膜が形成され細菌細胞が分裂する。枯草菌では複製開始点(oriC)から進む複製フォークの方向に75%の遺伝子が並んでいる15)。特に、高栄養条件下で転写 レベルの高いリボソームRNA遺伝子が複製の方向と逆向きになると、複製が途中で止まり、増殖できなくなるらしい16)。高栄養培地におけるダイズ根粒菌HRS株の生育速度が、non-HRS株と比較して遅くなる理由の一つとして、non-HRS株で整列化されていた複製と転写 の向きの関係がHRS株ゲノムでは崩れたことが考えられる。細菌ゲノムの遺伝子密度は高く、ゲノム進化の余地がないことは不思議であるとされてきたが、99%ともいわれる培養困難な土壌細菌のゲノムでは、複製と転写 の方向の整列化行われていないために、通常の培地で増殖しにくい可能性が考えられる。もしそうならば、大部分の土壌細菌は、ゲノム進化の壮大な実験場になっていることになる。
機能的に関連した細菌の遺伝子は、ゲノム上でひと固まりとなって、同じ転写単位として組織されている場合が多く、オペロンと呼ばれる。ゲノムの全塩基配列が決定された真正細菌と古細菌のオペロン構造を比較すると、オペロン内の遺伝子配列は進化の過程でかなりシャフルされているが、その乱雑度は、現在のゲノム中に存在している挿入配列(IS)のコピー数に比例している17)。ダイズ根粒菌HRS株は少なくとも200コピー以上のISを保有しているので、オペロン内の遺伝子配列は相当シャフルされているはずである。
3) 遺伝的多様性を生み出す変異はストレス条件下で多発する
HallやCairnsらは大腸菌をの栄養要求変異株の復帰変異という伝統的な細菌遺伝学の範囲で、増殖しないストレス条件下の細菌において復帰変異の頻度が高くなる現象を観察している18)。貧栄養条件下では、トランスポゾンなどが介在するゲノム再編成の頻度も高まることが知られている19)。また、複製フォークが転写と衝突すると、なんとか複製を続行させるために複製段階で遺伝子コピーの重複や欠失などの変異が生ずる15)。このような例を見ていくと、増殖していない細菌が貧栄養条件などのストレス環境下にさらされる場合に、遺伝的多様性が最も生み出されやすいのではないかと想像される。こういった背景から、ダイズ根粒菌HRS株も何らかのストレス要因によってISコピー数の増加とゲノムシャフリングが起こったものであると考えている。
4) フェロモンによる生存率のコントロールと休
眠状態からの蘇生
細菌はフェロモンによって集団的に生活環や行動の切り換えをおこなっている。高密度で培養されたインゲン根粒菌は定常期に入っても高い生存率を示すが、低密度で培養した菌の生存率は低下する20)。その原因は根粒菌が生産するホモセリンラクトンと言われる低分子化合物の濃度のしきい値によるものであった20)。また、胞子を作らないグラム陽性菌Micrococcus luteus の休眠細胞の蘇生に16~17 kDaのタンパク質性の蘇生促進因子(Resuscitation promoting factor, Rpfs)が関わっていることが最近明らかになった21)。
4. 地圏微生物のゲノム生態研究_統合的な土壌微生物研究の一つの方向性_
土壌細菌は、植物生態学などと比較すると確かに種類や現存量の把握は難しいが、逆に遺伝子がコンパクトにまとまってゲノムサイズが小さく、いくつかのモデル細菌ではその遺伝子発現・機能・それらのネットワークなどがかなり解明されつつある。そこで、逆にこのような利点を生かし、栄養飢餓、土壌粒子への接着、微生物間または植物との相互作用、環境中の物質循環への寄与などを研究して行くことは可能ではないだろうか。その際、ゲノム科学の論理ではなく、環境科学も包含した土壌微生物学の課題と論理で、環境食糧問題解決を目指しながら大きな共同研究プロジェクトで研究を行う必要があろう。
図4, 枯草菌の環状ゲノムの複製と遺伝子の転写方向
矢印は複製の方向を示し、円内の黒い部分は時計回りを向いている遺伝子の割合を示す。 oriC,
tetCは、それぞれ複製開始点と終止点を示す6)。
要旨
土壌細菌は地球レベルの物質循環や植物生育の上で大変重要な役割を果たしており、地球生態系の恒常性の維持に貢献している。本稿では、土着ダイズ根粒菌のDNAフィンガープリントを用いて、遺伝的多様性・風土性・ゲノムがシャッフルされたHRS株の遺伝生態研究について紹介を行った。土着ダイズ根粒菌も含めた土壌中の細菌の生活について細菌遺伝学やゲノム研究の最近の知見を踏まえて、_ゲノムの複製系と転写系の衝突、_オペロンの乱雑度と挿入配列、_非増殖条件による変異の誘発などについて考察を行った。今後、土壌微生物の分野でもゲノム研究、遺伝学分野、環境科学分野と連携し、総合性をもった研究が必要ではないだろうか。
謝辞
本研究を遂行する当たり、大部分の実験を行った伊沢剛博士と鮫島玲子氏に感謝します。また、本稿をまとめるに当たって貴重な助言を下さいました服部勉博士および三井久幸博士に謝意を表します。
引用文献