|
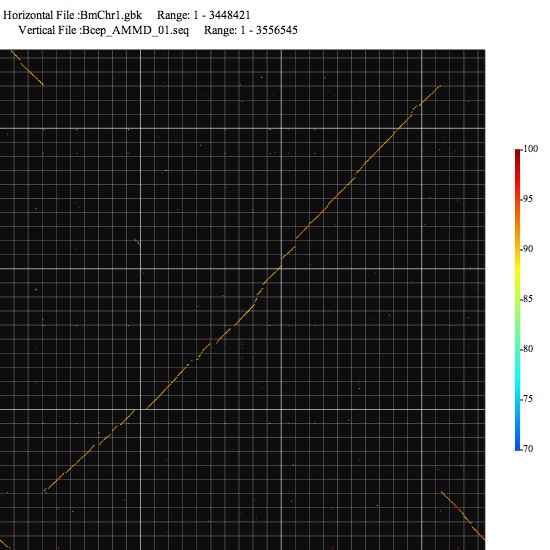
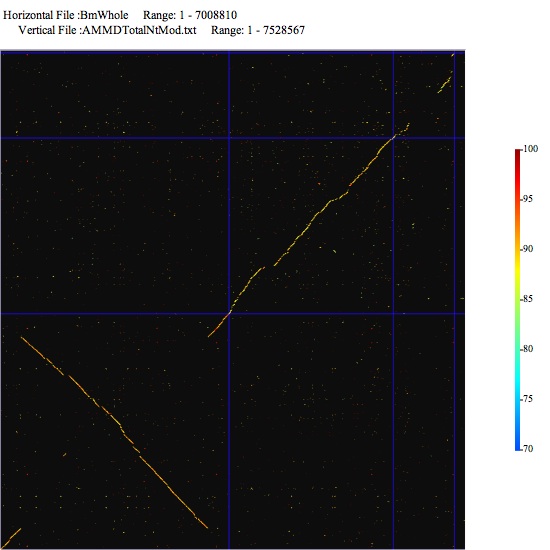
Image 1 画面の左下が原点。つづけて解析したい範囲をドラッグで選択し、さらなる解析を行うことができる。右側にあるカラースケールはidentityと色の関係を示している。カラースケールはゲノム間の相同性に応じて編集すると、相同性をつかみやすい。
 |
|
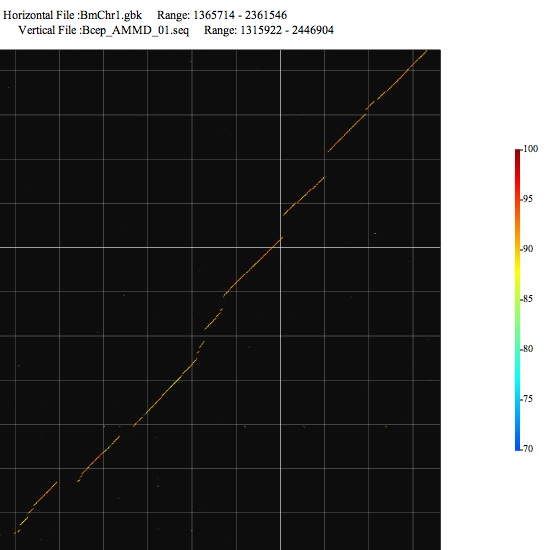
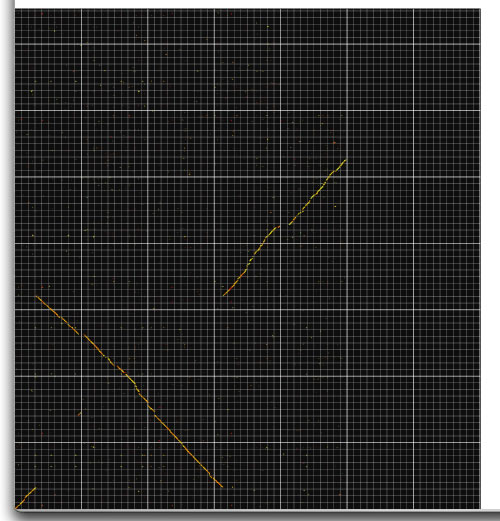
Image 2 上のimegeの一部分を再解析した結果。キーボードから解析範囲を入力することもできる。画像の任意の部分をシフトキーを押しながらドラッグすると平行移動する。
|
|
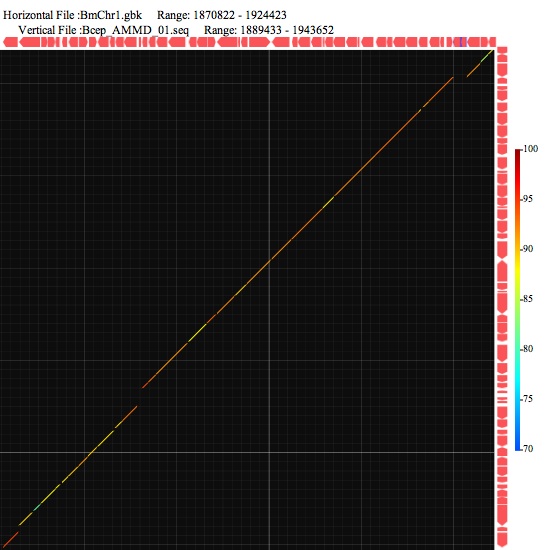
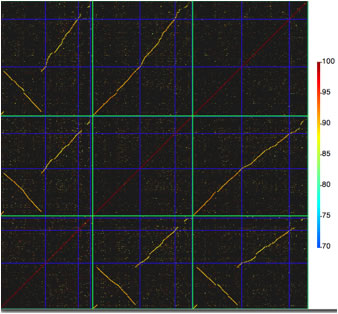
Image 3 上のimegeの一部分をさらに再解析した結果。周囲にある矢印は遺伝子を表すシンボル。コマンドキーを押しながらクリックすると色などをかえることができる。
|
|
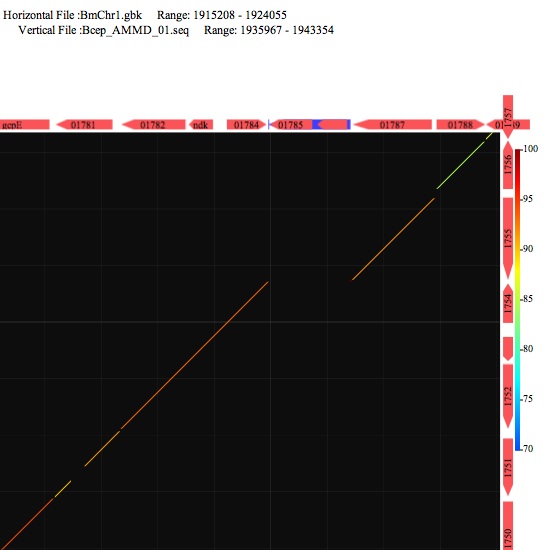
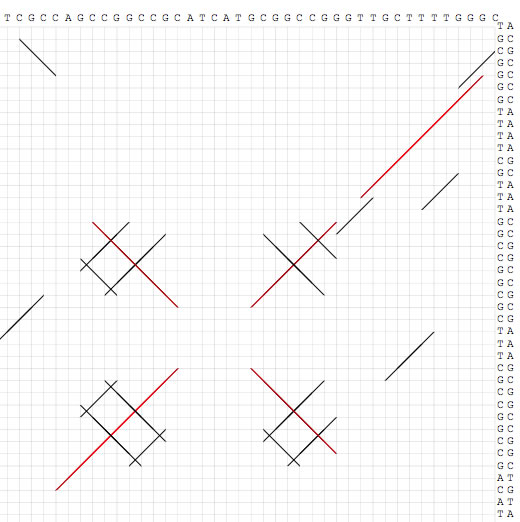
Image 4 上のimegeの一部分をさらに再解析した結果。クリックすると注釈情報が表示される。PDF形式で書き出せば、編集可能なグラフィックスを得ることが出来る。
|
|
Image 5 "show alignment by"プルダウンメニューの中から適当なプログラムを選択すると、選択範囲のアラインメントが表示されます。
|
|
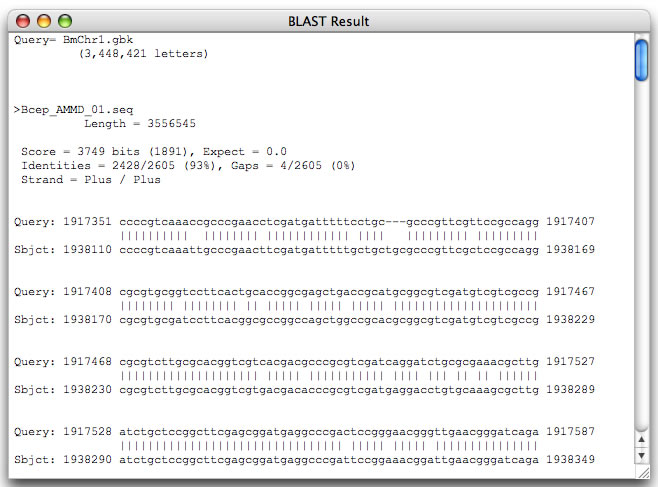
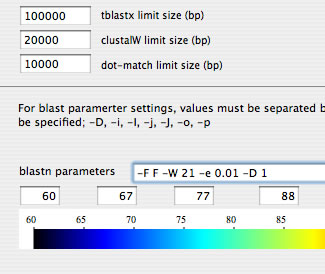
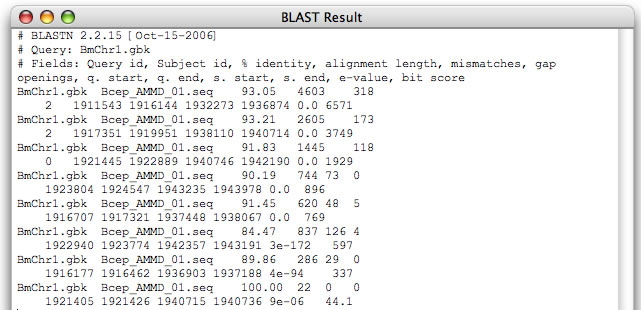
Image 6 セッティング画面で"-D"オプションに1をセットすると BLASTの結果をテーブルとして得ることが出来ます(表計算シートに貼り付けると良い).
|
|
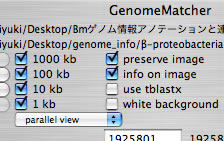
Image 7 パラレルビューを選択すると2つの配列は平行に配置される。下側の配列がx軸に読み込んだ配列。2次元比較図と同様にポインタで解析範囲を指定できます。この場合、画面の下側と上側別々にドラッグしてください。シフトキーを押しながらドラッグすると平行移動します。
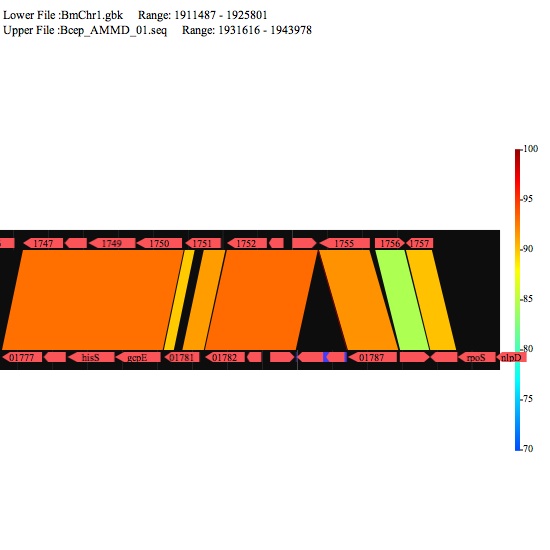
|
|
Image 8 "blastp"スイッチをオンにしておくとx軸上のCDSとy軸上のCDS間で総当たりでblastpがかかる。緑色の四角形がヒットがあったことを示している(数値はidentity)。 緑色の四角形をクリックするとアラインメントが表示されます。総当たりの結果は別のウィンドウにも表示されます(表計算シートに貼り付ける用のデータも得られます)。
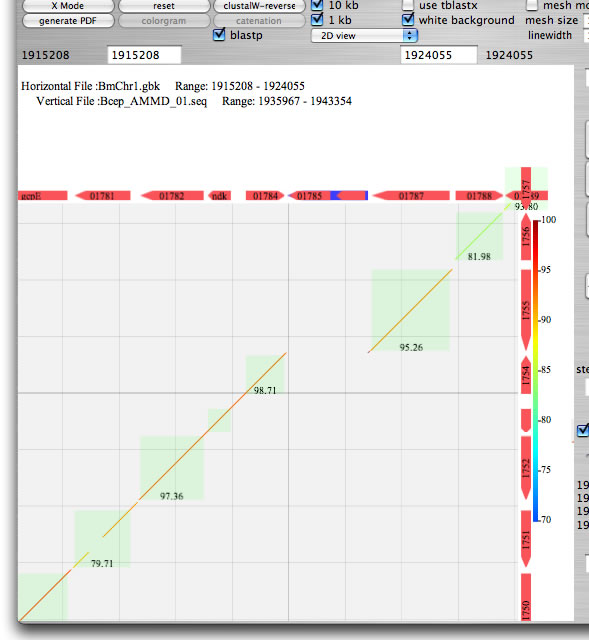
|
|
Image 9 アラインメントを得ることが出来る。ClustalW、MAFFT、bl2seq、MUMmerによるアライメントが取得可能です。MUMmer、MAFFTについてはユーザーのMacにこれらがインストールされている必要がある。
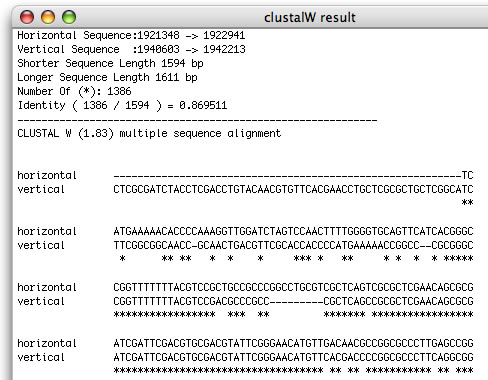
|
|
Image 10 カラーグラムによる2つの環状染色体の形の違いの表現。右側のテキストボックスにデータを入力することで直線と文字のセットを追加することができる。図は1度のinversionで説明がつく2つの染色体の構造の違いを表現している。12時が配列の開始点時計回り。
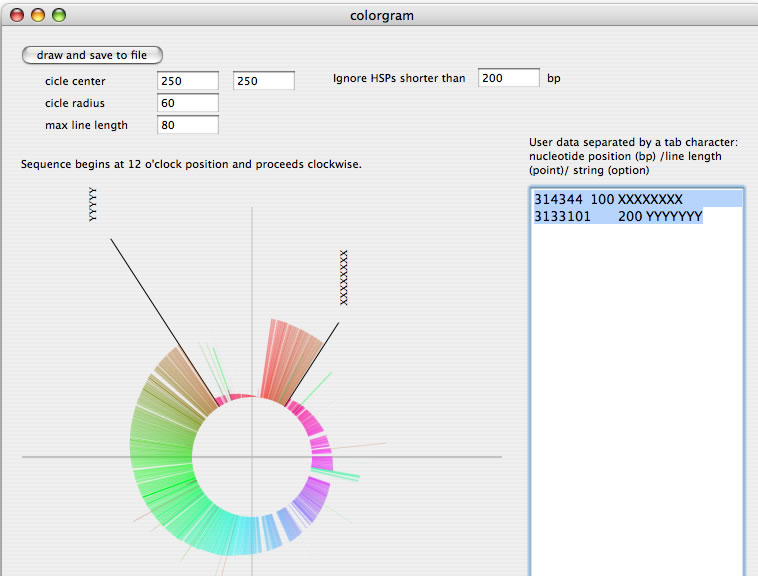
|
|
Image 11 マルチレプリコンの比較。レプリコンごとに順次比較される。レプリコンの境界を示す青い線は自動で引かれる。連結した配列は自分で作成する必要がある。version 1.25に連結した配列を作成する機能を追加しました。
|
|
Image 12 進行中のmeshモードのようす。指定した長さで区切られて順番に比較される。長大な配列でも短く区切ることで比較が可能。ここではmeshサイズを1Mにして実行。
|
|
Image 13 多ゲノムx多ゲノムの比較。それぞれのゲノムはマルチレプリコンより構成されている。ゲノムの境界は緑色で、レプリコンの境界は青色で示されている(色は選択可能)。サイズを指定することで好きな大きさの画像を得ることもできる。ポスターサイズの描画にも。
|
Image 14 ドットマッチによる解析の様子。見たい部位をドラッグで拡大できる。コマンド+Zでやり直し。解析画面のコピーをひらくことができる。

|
Image 15 比較範囲が100bp以内になると塩基が表示される。閾値を2つ設定することが出来る。1つめの閾値より長いマッチが黒で、2つ目の閾値より長いマッチが赤
|
Image 16 ドットマッチを使うと繰り返し配列の同定が容易。挿入配列の末端、複製開始地点、転写因子の結合部位、転写終結配列など生物学的に重要な部位の同定が容易になる。またPCR増幅しようとする領域に複雑な二次構造を取りうる部位があるかどうかを調べることが出来る。
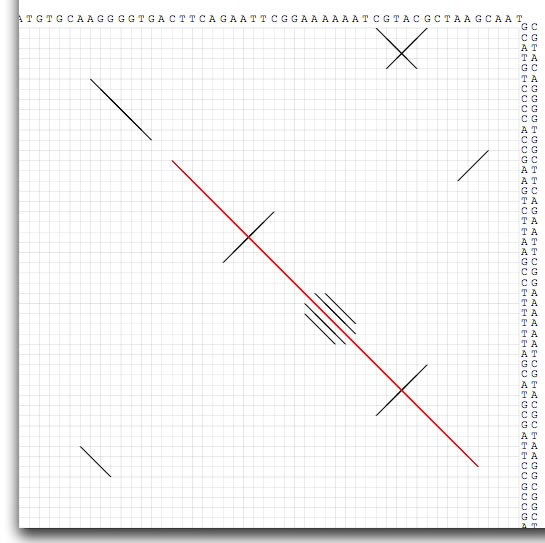
|
Image 17 シンテニー解析。設定した閾値以上の長さのギャップ部分が紫色で示されている。ギャップの位置をテキスト形式で得ることもできる。ISやゲノミックアイランドの位置を抽出するのに便利。抽出結果が正しいかどうかを目で見ながら判定できる。
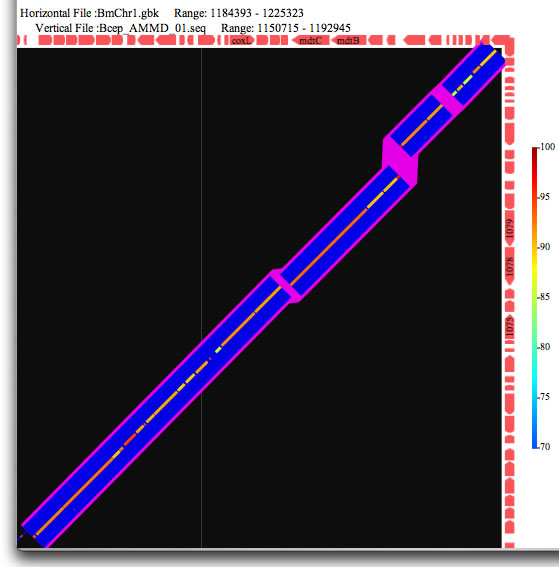
|
|
Image 18 Yモードによる解析。Yモードを使うことで離れた2箇所を近づけてみることが出来る。アイランドの末端形状の解析や、inversionの同定に。
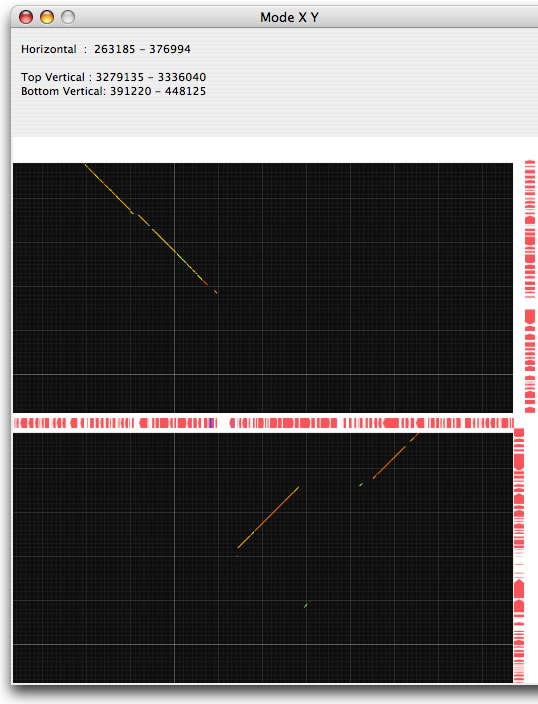
|
|
Image 19 外部配列をクエリにして相同性検索が実行可能。x軸の配列とy軸の配列両方に同時にblastがかかる。
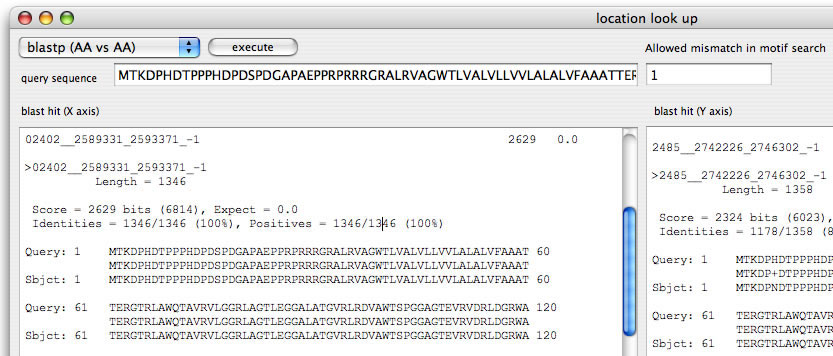
|
|
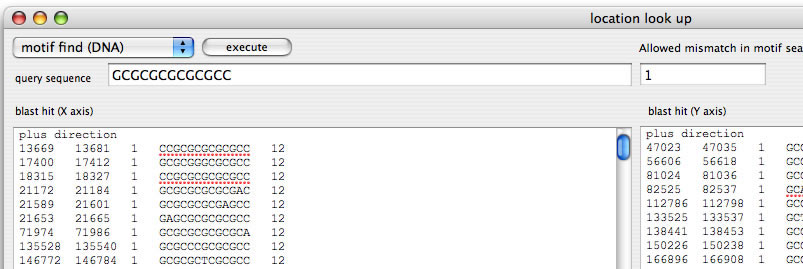
Image 20 モチーフサーチが実行可能。下の図はGCGCGCGCGCGCCと1塩基までの違いを許容してサーチを行った結果。結果は表計算シートに貼り付けると良い。
|
|
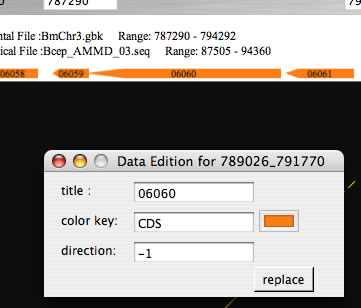
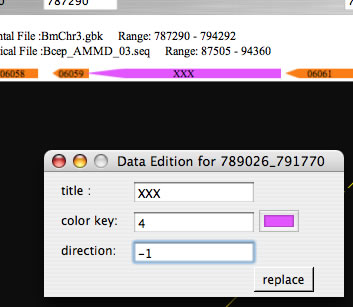
Image 21コマンドキーを押しながら矢印をクリックすると編集ウィンドウが開く。タイトル、色、向きを編集しreplaceボタンを押すと変更が反映される。
|
|
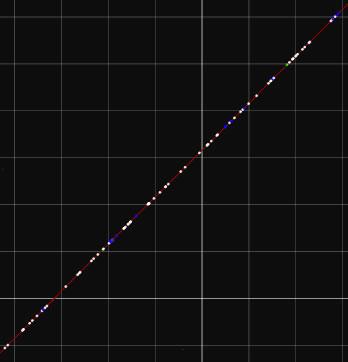
Image 22 SNP解析。MUMmerによるSNPの同定結果を、グラフィカルに図示できます。indelを青で、transitionを白で、transversionを緑で示しています。視覚的に点変異の位置を捉えることがます。結果をPDF出力できます(version 1.280以降)。
|
|
Image 23 blastpによる比較結果。ヒットが円で表されている。tblastnでx軸のCDSをクエリとしてy軸のDNA配列に対して、あるいはy軸のCDSをクエリとしてx軸のDNA配列に対して検索を実行できる(version1.280以降)。結果をPDF出力できる(version1.280以降)。
|
|
Image 24 表計算シートで、1行1エントリで管理されている情報をFASTA形式に変換する機能。「アクセサリ」→「StringFormatter」の一機能。
|
|
Image 25 CDS間でblastpをかけた結果をパラレルビューモードで表示。数字はidentity(%)。PDF出力もできる。
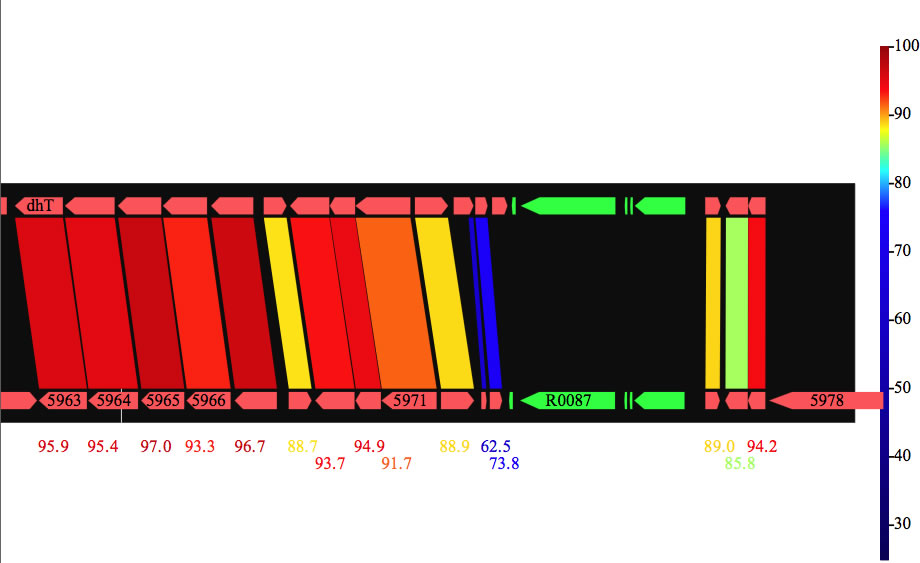
|
|
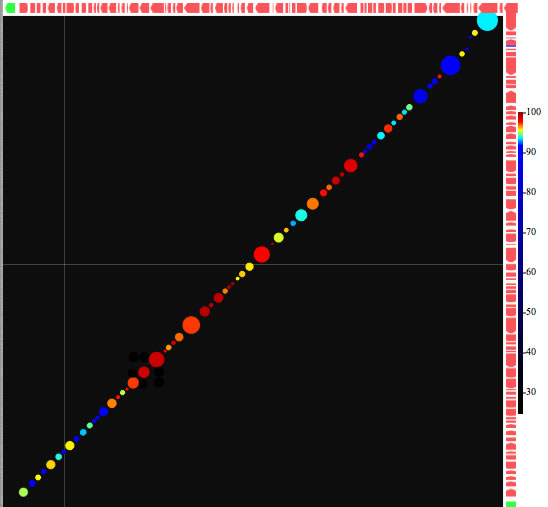
Image 26 相同性スコア(identity)の分布図。しきい値を設定すると、しきい値以上のidentityを持つヒットについてのみを表示できる。また分布図上でコントロール+クリックで相同性分布データを参照できる。
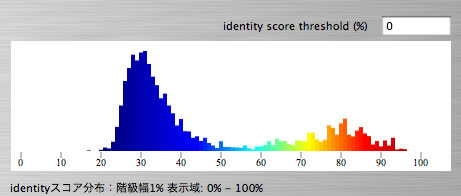
|
|
Image 27 付属機能「ExtractFromGenBankFile」の実行結果。GenBankファイルの内容を表計算シートで取り扱いやすい形式に変換する。どのデータを出力するかを指定可能。
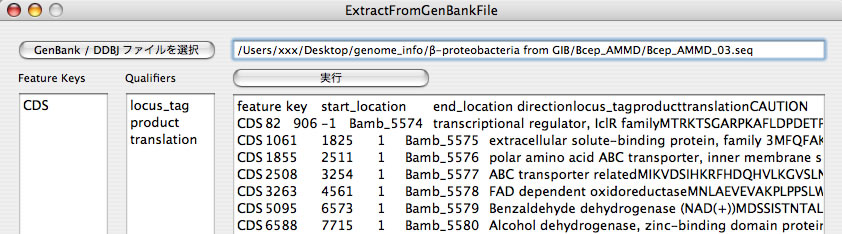
|
|
Image 28
1対1の比較を繰り返すモードを付けました。コマンド+2でwindowが開きます。このモードから総当たり比較もできます。あらかじめ興味のある遺伝子に色を付けておき、遺伝子をコントロール+クリック
し、「set identical color to similar CDS」を選択すると、相同性のある遺伝子に同じ色をつけることが出来ます。色を付けた遺伝子がたくさんあるときはパネルをコントロール+クリックしてsynchronize color for similar CDSsを選択してください。相同性のしきい値、クエリの何%以上相同性があるときに似ていると判断するかどうかは、コマンド+Bのwindowで設定できます。

|
|
Image 29
複数の配列を比較する機能に、特定の遺伝子にフォーカスする機能を付けました(コンテクストメニューからseach and focusを実行)。アミノ酸配列あるいはDNA配列をクエリとして各パネルに読み込んだ配列に対して相同性検索を実行し、トップヒットの遺伝子が中央に来るように描画されます(2番目、3番目のヒットが中央にくるようにすることもできます)。
またヒットした遺伝子に指定色をセットすることができます。
|
|
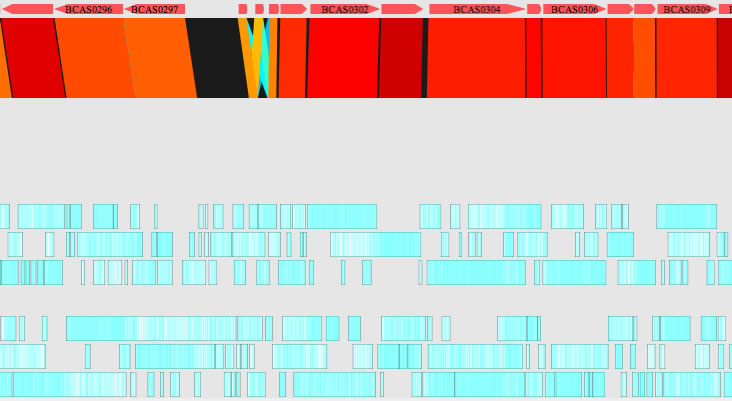
Image 30
アノテーション支援パネル。ORFを6フレームについて抜き出します。図では上のパネルに読み込んだ類縁配列のCDSをクエリとしてtblastnをかけた結果を示しています。各ORFの各コドンの3番目がGかCである部分を青色で示す機能がついています。
GC含量が高い生物ではコドンの3番目のGC含量が他のフレームと比べて高いので、どのORFがCDSであるかをある程度判定することができます。
|
|
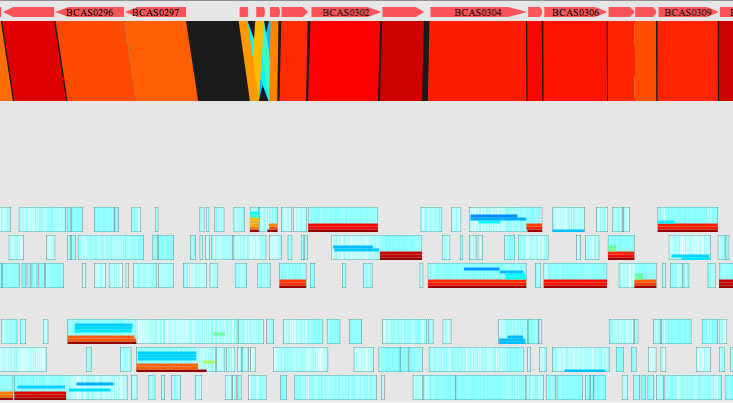
Image 31
Image30の全ORFについてblastpで解析した結果を示します。この結果をみると、どのORFのどこからどこまでがデーターベース中の配列とどのくらいの相同性があるかを把握することができます。
さらに.....(image32を見てください).
|
|
Image 32
さらにCDSであると推定されたORFをコマンドキーを押しながら上方向にドラッグすると、開始コドン位置決定用のwindow(image 33)にORFが登録されます。登録されているORFはやや細い長方形として表示されています。
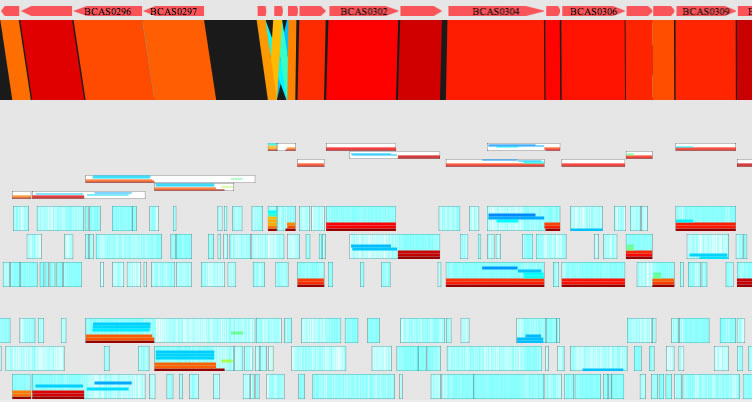
|
|
Image 33
開始コドン決定用のwindowの様子。上の矢印は、リファレンス配列中のもっとも高い相同性を示したCDS。縦の線は開始コドンとなりうるコドン(ATG、GTG、TTG)の位置を示します。赤線は開始コドンとして登録されているコドン。下の矢印は開始コドンを決定しようとしているORF。上の矢印の横一直線のラインは
、下の矢印と相同性のある部分。下の矢印のラインは、
データーベース中の配列と相同性が見られた部分。図のようにリファレンスとなる配列の開始コドンとその上流の配列を見ながら、開始コドンの位置を選択することができます。なお、描画レートを8000 point / kbにするとORFの塩基配列を表示することができます(image 34)。 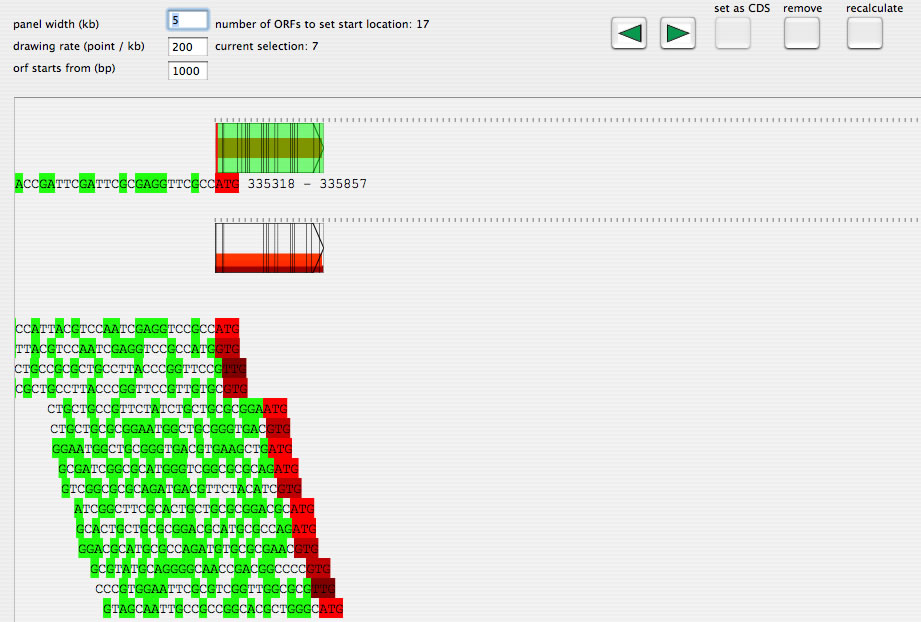
|
|
Image 34
開始コドン決定用のwindowで描画レートを8000 point / kbにするとORFの塩基配列を表示することができます。これと思う開始コドンを選択して「set as CDS」ボタンを押すとCDSとして登録されます。
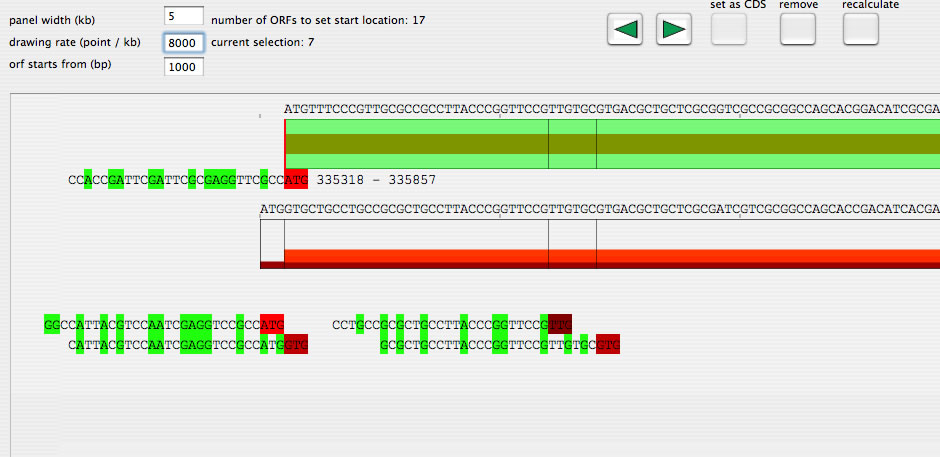
|
| |




































