この機能は付属機能である「CompareSequences」の一機能です。DNA配列の中のタンパク質をコードしている部分(CDS)を同定し、その開始コドンがどこであるかを効率良く決定することができます。また最新バージョンでは、異なるオートアノテーションシステム由来の2つの注釈ファイルを読み込んで、差のあるところを重点的にチェックする機能、一括変更が容易な表計算シート形式で注釈データを出力する機能が追加されています。なお、表計算シート形式は、編集後、StringFormatter機能を使用することでDDBJのマスサブミッションツールに適合する形式、NCBIのtbl2asnが読み込めるtbl形式に変換することができます。
はじめに
遺伝研のMiGAPやNCBIのPGAPといったオートアノテーションシステムによるCDSの予想開始点は信頼性に欠けるため、開始コドンの位置を1つずつ確認/修正する必要があります。開始コドンが適切かどうか判定し、必要に応じて修正する作業はかなり大変です。GenomeMatcherのアノテーション支援機能は、CDSの開始コドンを手作業で決定するのを支援する目的で作られたものです。注釈無しのDNA配列中のCDSを同定することができますが、オートアノテーションのCDSの開始点について解析し、変更することができます。
手作業で頭決めを行う場合、一般的に参考にされているのは、(1)開始コドン直上流にSD配列らしきものがあるか、(2)近隣のCDSとの位置関係、(3)データベース中のアミノ酸配列と似ているところはどこか、(4)類縁ゲノムでどの部分がCDSとされているか、であるかと思います。本支援機能では、これらデータを同時に俯瞰し、適切な開始コドンを素早く選択することができます。また解析に時間がかかる部分はまとめて処理できるようになっており、数秒単位の待ち時間が発生しないようになっています(数秒程度の待ち時間が繰り返されると、集中力が低下してつらい...)。作業結果は、表計算シートに適合する形式で出力されます。必要に応じてデータを編集し、StringFormatterを利用してデータバンクが受け付ける形式に変換してください。
アノテーションパネル
アノテーションパネルを追加するには「add annotation panel」ボタンを押します。アノテーションパネルは1つしか作ることができず、また全てのパネルの一番下に配置されます。
「file」ボタンからDNA配列を含むファイルを選択します(選択すると「file」ボタンが「draw」ボタンに変化します)。
通常のパネルと同様に、上のパネルと比較する範囲の開始位置、終了位置、向き、配列のニックネーム、オフセットを指定します。また通常のパネルに加えて以下のテキストフィールドやボタンがあります。
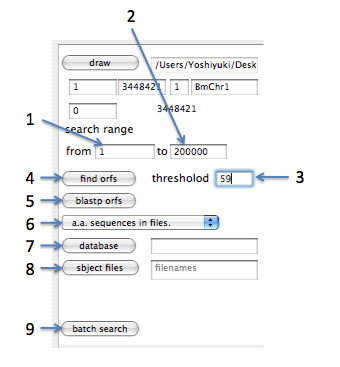
- ORF抽出する範囲およびblastpにかけるORFの開始位置(図の1)、終了位置(図の2)を指定するためのテキストフィールド
- 抽出するORFのサイズのしきい値を指定するためのテキストフィールド(図の3)
- ORFを抽出するためのボタン「find orfs」ボタン(図の4)
- 指定範囲にあるORFをblastpにかけるための「blastp orfs」ボタン(図の5)
- 既存のBLASTデータベースを利用するのか、あるいは、ファイル中のアミノ酸配列をデータベース化して利用するのかを選択するプルダウンメニュー(図の6)
- BLASTデータベースを指定するための「database」ボタン(図の7)
- ファイル中のアミノ酸配列をデータベース化して利用するとき、ファイルを選択するための「sbject files」ボタン(図の8)
- 様々な解析を実行するためのwindowを開くための「batch search」ボタン(図の9)
ORFの抽出と消去
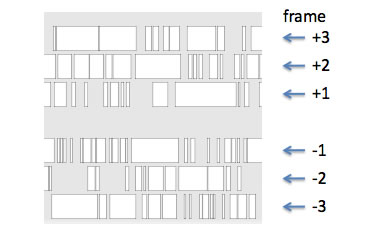 ORFを抽出する範囲を指定します(テキストフィールドに数値を入力するか、アノテーションパネル上をドラッグします)。抽出するORFの長さについて適当なしきい値を指定したら「find orfs」ボタンを押します。6行に渡って白い四角形が現れます。上からFrame +3、+2、+1、-1、-2、-3です。
ORFを抽出する範囲を指定します(テキストフィールドに数値を入力するか、アノテーションパネル上をドラッグします)。抽出するORFの長さについて適当なしきい値を指定したら「find orfs」ボタンを押します。6行に渡って白い四角形が現れます。上からFrame +3、+2、+1、-1、-2、-3です。
ORFを消去するには、各ORFのコンテクストメニューから「delete」を選択するか、アノテーションパネル上で消去したいorfsをドラッグして囲み、アノテーションパネルのコンテクストメニューから「delete orfs in dragged range」を選択します(薄緑色の選択範囲に完全に含まれるorfが消去されます)。
ORFのblastp解析
- どの範囲に存在するORFをblastpにかけるかを指定します。
- プルダウンメニューから既存のBLASTデータベースを利用するか、ファイル中のアミノ酸配列をデータベース化して利用するかを選択します。
- 前者の場合、「database」ボタンを押してデータベースを選択します。後者の場合、「sbject files」ボタンを押してアミノ酸配列を含むファイルを選択します(複数可、multi FASTAまたはGenBank形式のファイル)。
- 「blastp orfs」ボタンを押します。
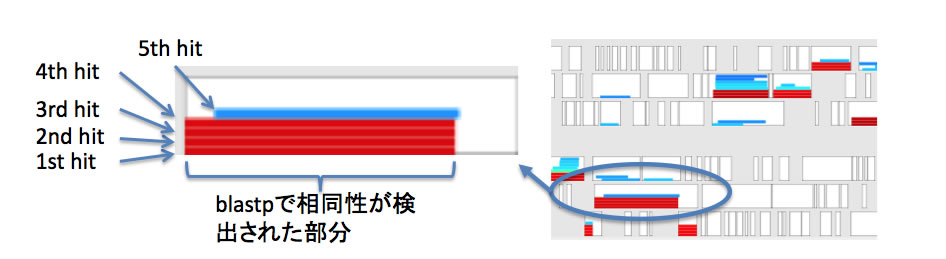
- ORFのどの位置がblastpでヒットしたかが、白いORF上に最大8件まで線で表示されます。このとき、色は相同性を、線の位置がヒット位置を表します。スコアが高いヒットが下にくるようになっています。
コドンの3rdポジション
 「display 3rd position GC in annotation panel」スイッチをオンにしておくと、各ORFについてコドンの3番目がGあるいはCである場所に青い線がひかれます。ORFにCDSが含まれるかどうか、あるいはORFのどこからがCDSであるかを判定するのに役立つかもしれません。このスイッチをオンにすると非常に重くなるので気をつけて下さい。
「display 3rd position GC in annotation panel」スイッチをオンにしておくと、各ORFについてコドンの3番目がGあるいはCである場所に青い線がひかれます。ORFにCDSが含まれるかどうか、あるいはORFのどこからがCDSであるかを判定するのに役立つかもしれません。このスイッチをオンにすると非常に重くなるので気をつけて下さい。
バッチサーチ
CDSを取りこぼしていることが推定される部位(CDS間が長い部位)が複数ある場合に、これらを一括して解析するための機能です。
「batch search」ボタンを押して開くwindow中の機能を利用すると、DNA配列中の複数の範囲についてORF抽出とblastp検索を、一括して実行できます。またそれぞれの範囲へとジャンプするボタンが作成されますので結果を確認する際に便利です。このボタンは、最後に押したボタンに赤枠が付きます。またシフトを押しながらクリックすると網掛けになり、もう一度押すと網掛けが解除されます。
他の配列との比較解析
参照配列があれば、他のパネルに読み込んでおきます。一つ上のパネルで比較プログラムとして5番を指定してcompareボタンを押すと上のパネルでアノテートされているCDSをクエリ、アノテーションパネルのDNA配列をデータベースとしてtblastnが実行され結果が表示されます。
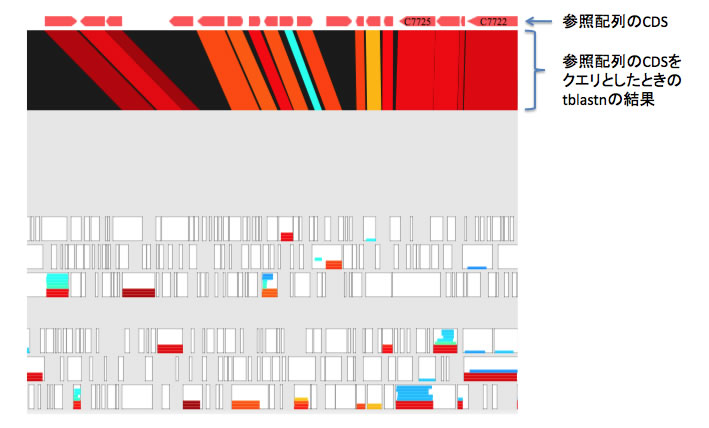
開始コドン決定用windowへのORFの登録
参照配列がある場合は、他のパネルで読み込んでおきます(複数可)。
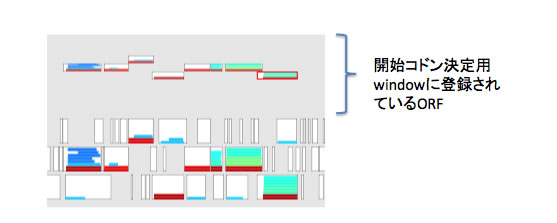
対象ORFを開始コドンの位置決定用のwindowに登録します。ORFを表す四角形を、コマンドを押しながら上方向にドラッグすると(わずかにドラッグするだけでOK)登録されます。
既に存在しているCDSを、開始コドン決定用windowに登録するには、対象となるCDSが含まれるようにその範囲をドラッグした後に、アノテーションパネルのコンテクストメニューから、「register CDSs in dragged range to start location identification window」を選択します。
登録されたORFは細い四角形として6フレームのORFの上部に表示されます。
開始コドンの位置決定用window中の情報の見方
ORFを登録するとそのORFのアミノ酸配列をクエリとして、アノテーションパネル以外の全てのパネルに対してblastpがかけられます。各パネルについてトップヒットしたCDSが、表示されます。図に例を示します(クリックすると拡大します)。
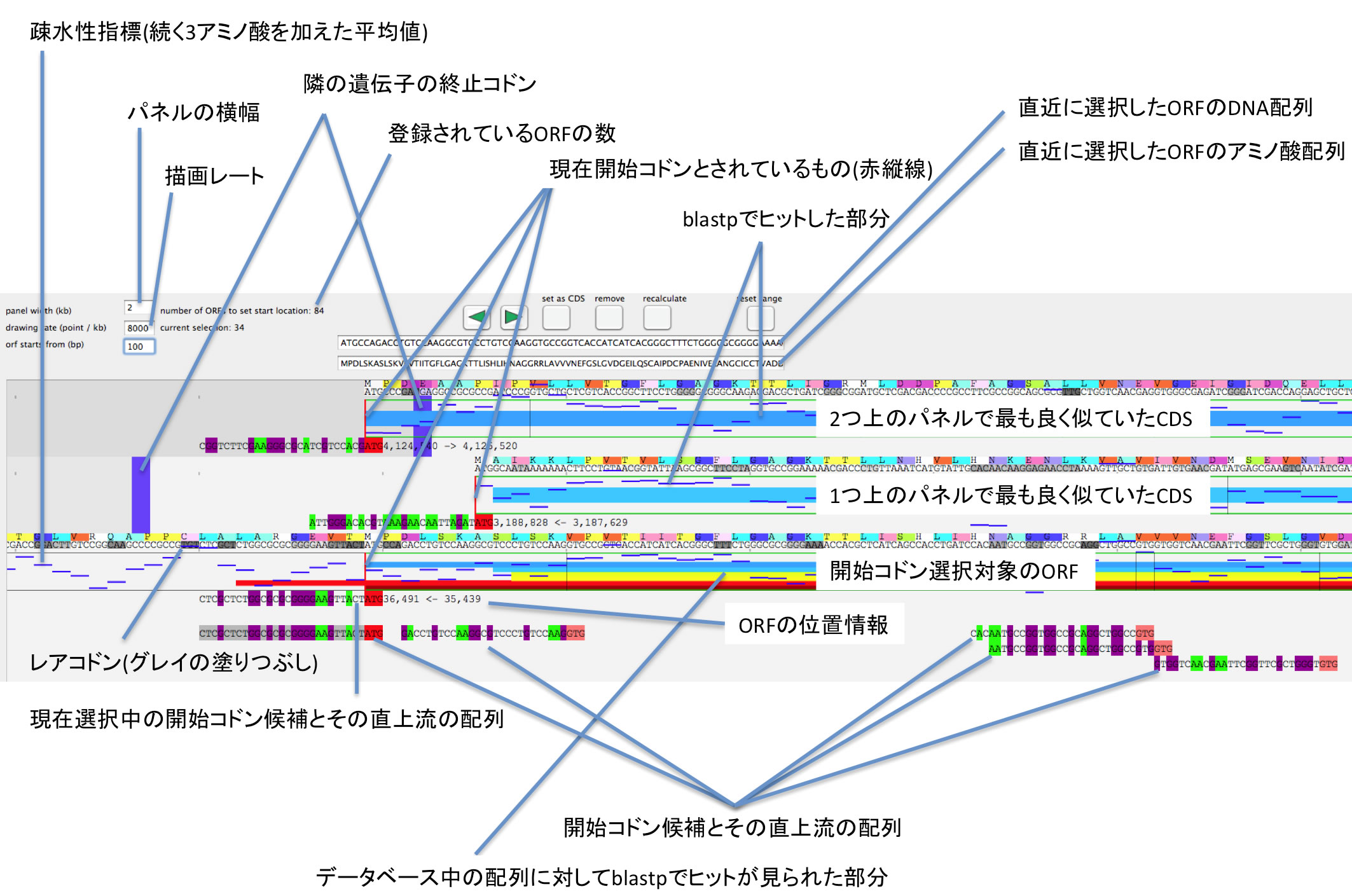
- 一番下にあるのが、開始コドンを決めようとしているORFです。
- 上にあるのは、参照配列の中で最も似ていたCDSが含まれるORFです。
- 縦線は開始コドンとなりうるトリプレットの位置を示します。
- 赤い縦線は、現在開始コドンとされているトリプレットです。
- 矢印内部をクリックすると、開始コドントリプレットが選択され、その上流30塩基が表示されます。トリプレットの一文字目の左側が縦線の位置になります。また開始コドントリプレットから終止コドンまでの間が薄緑色になります。
- 上のパネル中の太い横線は、これから開始コドンを決めようとしているORFのアミノ酸配列をクエリとしてblastpをかけたときに検出された相同部位です。
- これから開始コドンを決めようとしているORF中の横線は、先にblastpをかけたときの結果です。
- 一番下にあるのが、各開始コドン候補の配列です。
- 水色の横線は、アミノ酸残基の疎水性の指標です。そのアミノ酸と続く3つの計4つのアミノ酸残基の疎水精度の平均がプロットされています。
- DNA配列が灰色に塗られている箇所は、アノテーションパネル中のCDSのコドン使用頻度から見たときのレアコドンです。灰色が濃いほどレアです。
- DNA配列の上にある色付きの四角は、アミノ酸配列を色で表しています。
- パネルの幅は、kb単位で設定できます。
- 1kbを何ポイントで描画するか指定します。8000 point / kbとすると特別な効果を得ることができます(DNA配列が表示されます)
- ORFの位置情報が表示されています。
クリックすると拡大します。
開始コドンの位置決定用window中の操作
開始コドンを選択して「set as CDS」ボタンを押すと、CDSが新規に登録あるいは既存CDSの開始コドンが変更されます。新しく登録したCDSにはカラーコードとして「newCDS」が、開始コドンを変更したCDSには「newStart」が設定されます。
開始コドン決定用windowから除くには、「remove」ボタンを押します。
参照ゲノムに対するblastp解析を再度行うには「recaluculate」ボタンを押します。新たにパネルを追加したときなどにお使い下さい。
左矢印ボタン、あるいは右矢印ボタンを押すと、登録されている他のORFを表示します。現在表示中のORFはアノテーションパネル中で赤枠がつきます。
登録されているORFの数と現在何番目のORFを表示しているかが表示されています。
直近に選択されたORFのDNA配列とアミノ酸配列が出力されています。
オートアノテーションによるCDSの開始コドン位置の見直し
オートアノテーションで同定されたCDSについて、開始コドンの取り方を変更するやり方について述べます。まずは、1つの注釈付きDNA配列について全てのCDSの開始位置を見直す場合です。
1: 見直したいCDSの、stopコドンの位置を表計算シート上で編集します。
 (コピーペースとして使います)
(コピーペースとして使います)
2: パネルを追加して、リファレンスゲノムを(必要に応じて複数を)読み込みます。また、異なるアノテーションシステムによりアノテートされたデータがあれば、同様に読み込んでおきます。
3: アノテーションパネルを追加して、対象となる注釈付きDNA配列を含むファイルを読み込みます。
4: 解析対象範囲として、配列の全体を指定した上で、「find orfs」ボタンを押します。
5: blastp検索に用いるデータベースのソースをプルダウンメニューから選択します。必要に応じてblastp実施可能なデータベースを選択するか、アミノ酸配列が含まれるファイルを選択します。
6: 「batch search」ボタンを押してウィンドウを開き、1で編集したデータを貼付けます。
7: テキストボックス右上の「orfs」ボタンを押します。4で見つかったORFのうち、1で編集した終止コドン位置と一致するものが、開始コドン位置決定対象として解析が始まります。最初は10個程度のデータを指定して、解析にかかる時間を見積もってください(nrに対して1000件のblastpを実施すると、数時間はかかるかと思います)。
8: ひたすら開始コドンを見直します。開始コドンを選択→「set as CDS」ボタンを押す→「remove」ボタンを押す→次のCDS。後いくつ残っているかは画面のやや左の上部に表示されていますので励みにしてください。
9:開始コドンを見直し終わったらデータを取り出します。「batch search」ボタンを押して開くwindow中でexportボタンを押してください。ローカスタグを付け直したい場合は、チェックボックスにチェックをいれ、ローカスタグを何番からつけるかを指定してください。ローカスタグは5番ずつ増加する様になっています。変更に伴ってprotein_idも更新されます。
続いて、1つのファイルについて異なるアノテーションシステム由来のファイルがある場合に、開始コドンが異なるもののみを見直し対象とする場合です。この場合、アノテーションパネルには、ベースとするアノテーションファイルを読み込んでください。また、他のパネルに参照とするアノテーションパネルを読み込んでください。blastp用のデータベースを選択し、「batch search」ボタンを押して開くwindow中で「analyze」ボタンを押してください。データの取り出し方などは、上述の通りです。
アノテーションデータの処理
exportしたデータは、表計算シートに貼付けます。color_keyでソートすると新たに追加したCDS、開始コドンの位置を変更したCDSを集めることができます。新たに追加したCDSについては、product名を決める必要があります。(1) 一つ一つNCBIなどでblastpサーチをかけてproduct名を決める、(2) KAASを利用して決める、(3)参照になるオートアノテーションファイルがあればそのproduct名をつける、などしてproduct名を決定してください。noteなどを追加するなどして、表計算シートのデータが完成したら、CAUTION列、color_key列を除く部分を、NCBIに投稿するのであればtbl形式、DDBJに投稿するのであれば、DDBJのマスサブミッションツールが要求する形式に変更します(StringFormatterでできます)。その後の処理については、各データバンクのデータサブミッションのページを参照してください。
ヒントと注意
blastpでデーターベースにnrを指定すると1つのORFに30秒程度はかかります。長大なDNA配列からCDSを同定する場合は、類縁種のアミノ酸配列のデーターベースを用意してblastp解析を行い、相同性が見られなかった部位に対してのみnrなどの大きなデーターベースを利用すると良いでしょう。
BLASTデーターベースの作成を支援する機能がMassiveBLASTにあります。「ファイル中のアミノ酸配列をデータベース化して利用する」場合、blastpを実行するたびにファイルを読み直しますので時間が余計にかかります<対応予定>。
ORFを探すときは、選択範囲に全長が含まれるORFが抽出されます。
ORFボタンはクリックできます。
1つのORFを複数回blastpにかけたとき、おなじsbject nameに関するヒットは2度目以降、排除されます。