CompareSequences
複数の配列について1対1の比較を繰り返して表示する機能です。つまり配列Aを配列Bと、配列Bを配列Cというように順番に比較することができます。複数の配列それぞれに対して、興味ある遺伝子の相同遺伝子を探して自動的に表示位置を調整する機能がついています。この機能を使うと興味ある遺伝子の周辺構造について、例えば10種の近縁種について相互比較することができます。また総当たり比較をするモードへ移行することができます。

アノテーション支援機能を利用できます。この支援機能は開始コドンの位置決定に特化していますので、開始コドンの位置決定に集中して取り組むことができます。
はじめに
いくつかの遺伝子よりなる遺伝子クラスターに興味を持っており、これが類縁ゲノムの中でどのようになっているか調べたいことがあるかと思います。ゲノムの一部分について相互に比較したいような時は、CompareSequencesをご利用ください。また「CompareSequences」には、他の配列のアノテーションを参照しながら遺伝子の頭(開始コドン)を効率良く決定/修正できるように工夫したアノテーション支援機能がついています。複数のゲノムについてその全体構造を比較するにはメイン機能の「catenation」機能が便利です。
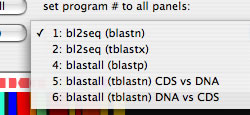
本機能では、各プログラムに番号を割り振っておりその番号でプログラムを指定します。番号とプログラムの関係は以下の通りです。
- 1: bl2seqのblastn
- 2: bl2seqのtblastx
- 4: blastallのblastp
- 5: blastallのtblastn。ただし、番号を指定するパネルのCDSを、下にあるパネルのDNAを比較。
- 6: blastallのtblastn。ただし、番号を指定するパネルのDNAを、下にあるパネルのCDSと比較。
新しいパネルを追加するには「add empty panel」ボタンを押します。各パネルには1つの塩基配列を読み込むことができます。各パネルについて、以下の設定をします。
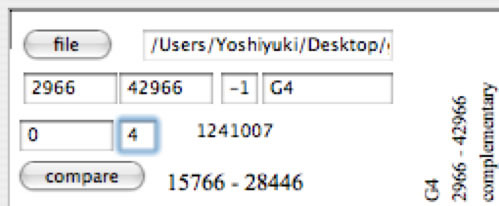
- 比較開始位置と終了位置(下図で2966 - 42966)
- その部分をパネルの左側、あるいは右側を若い位置番号とするか(1:左側、-1:右側: 下図では-1)
- その配列のニックネーム(任意の名前のこと:下図でG4)
- オフセット(左側を何ポイント空けるか: 下図で0))
- 比較プログラム番号(下図で4: blastp)(ここで指定したプログラムで、このパネルと一つ下にあるパネルが比較されます)。
 コントロールパネル。
コントロールパネル。
なお1241007は配列の全長。15766-28446はドラッグした範囲。縦書きになっているのは左から、ニックネーム、現在表示されているイメージの比較範囲、配列の向き(ここではcomplementary(-1))。
パネルの幅と描画サイズ
パネルの幅と描画レートを指定します。パネル幅を200kb、描画レートを20ポイント/kbとすればパネルの幅は4000ポイントとなります。解析の比較対象範囲がパネル幅より長い場合、はみ出した部分については解析はされますが表示されません。
逆にパネルの幅が100 kbだとしても、比較開始位置と終了位置の間が10 kbであれば、その10 kbしか表示されません。
簡単な使い方
- パネルを何枚か追加して、各パネルにDNA配列を読み込みます(「file」ボタンを押してファイルを選択)。
- 興味ある遺伝子の配列(アミノ酸配列あるいはDNA配列)を用意します。ここではアミノ酸配列とします。
- どれか1枚のパネルのコンテクストメニューから「search and focus」を選びます。
- 開いたwindowのqueryテキストフィールドに配列をペーストします。
- 検索するのに利用するプログラムを「search program」プルダウンメニューから選択します。ここではblastpを選択します。
- 見つかった遺伝子にカラーコード「blue」を指定することにします。
- 「run (all panels)」ボタンを押します。
入力したアミノ酸配列をクエリとして、各パネルの配列中のアミノ酸配列をBLASTデータベースとして、blastpが実行されます。トップヒットのCDSがパネル中央に右向きに配置されるように解析位置範囲と配列の向きが設定されます。また各パネルで指定のプログラムで下にあるパネルの配列と比較されて結果が表示されます。
解析範囲を変更する
解析範囲を変更するにはいくつかの方法があります。
- コントロールパネルで開始位置、終了位置を手入力する。
- パネルをシフトキーを押しながらドラッグする。
- 解析したい位置をドラッグで選んでから、パネルのコンテクストメニューから「set drag range」を選択する。
比較プログラムを変更する
プルダウンメニューからプログラムを選択すると、全てのパネルの比較プログラムがここで選択したプログラム番号に変更されます。メニュー中に数字とプログラムの対応関係が示されています。また各パネルのコントロールパネルで、比較プログラムを数字で指定することもできます。
 プルダウンメニュー
プルダウンメニュー
解析条件の保存と復元
解析条件を保存/復元する方法は2つあります。1つは、読み込んでいる配列へのパスや解析範囲、解析プログラムなどを文字情報として出力し、それを表計算シートで保存する方法、もう一つは、現在の解析情報について専用のファイルを作って保存する方法です。いずれの場合にも、復元時には既に存在しているパネルはそのままに、復元されたパネルが追加されます。
前者については「import / export settings」ボタンを押して専用のwindowを開き、そこで「export settings」ボタンを押します。各パネルのコントロールパネルに入力されている値がタブ区切りで出力されます。パネルを復元するには、テキストフィールドにデータを貼り付けて「import settings」ボタンを押してください。このとき、パネルの順番、解析位置などをお好みに併せて編集することができます。出力形式と入力形式は同一です。
 出力の例
出力の例
後者は、「save panels」ボタンを押して、現在の解析データを保存します。復元するには「load」panlesボタンを押し、次いでファイルを選択します。復元時にアノテーションパネルが既に存在している場合は、選択したファイルにアノテーションパネルが含まれていても復元されません。
パネルを編集する
パネルが沢山あるとき、パネルの順番を変えたり、複製を作ったり、パネルを削除したりといった操作を簡単に実行する機能があります。「control panels」ボタンを押して専用のwindowを開いてください。詳しいヒントが画面上に表示されていますので参照して下さい。
また各パネルをコマンドを押しながらドラッグすることでパネルの順番を変更できます。ドラッグしているパネルが挿入される位置は赤い線で示されます。
パネル間を比較する
各パネルのコントロールパネルには「compare」ボタンがあります。これを押すとそのパネルとその下にあるパネルの比較が実行されます。また「compare & draw all」ボタンを押すと全てのパネルの「compare」ボタンを押すのと同等の効果があります。「caluculate immediately with new neighbors」スイッチをオンにしておくと、ドラッグ直後に再解析、再描画が行われます。
search and focus機能を使う
「search and focus」は興味ある配列の相同配列がどこにあるのか探して、焦点をあてる機能です。パネルのコンテクストメニューから「search and focus」を選択すると専用のwindowが開きます。見いだされた配列がパネル中央に右向きになるように、解析範囲と向きが変更されます。
アミノ酸配列あるいはDNA配列をクエリとすることができます。検索に用いるプログラムをプルダウンメニューから選択してください。blastpを選択した場合は、見つかった配列に任意のカラーコード(下図ではblue)を指定することができます。
トップヒットではなく2ndヒットに焦点をあてたいときはwindow左下のテキストボックスに2を入力してください(図では1になっている)。
「run (single panel)」ボタンからfocus機能を実行すると、コンテクストメニューの呼び出しもとのパネルに対してのみ実行します。
検索結果は順次、下にあるテキストフィールドに出力されます。multi FASTA形式でも出力されます。
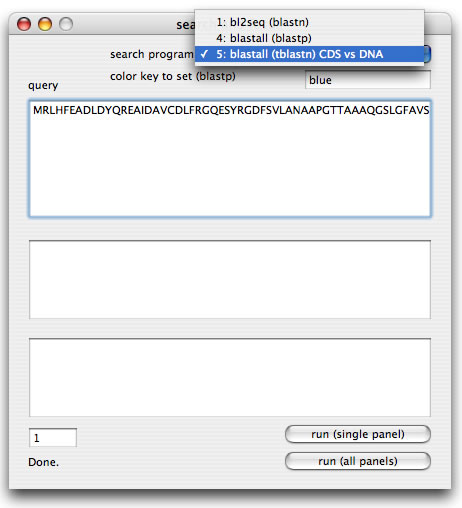
相同遺伝子に同じ色を付ける
相同遺伝子に同じ色を付けるには2つの方法があります。1つは遺伝子シンボルのコンテクストメニューから「set identical color to similar CDSs」ボタンを選びます。この場合、この遺伝子に付けられているカラーコードを、相同遺伝子にセットすることができます。もう一つは、各パネルのコンテクストメニューから「synchronize color for similar CDSs」を選択する方法です(下図)。この場合、コンテクストメニューを呼び出したパネルの解析範囲にある全てのCDSについて、他のパネルの解析範囲にある相同遺伝子に同じカラーコードをセットします。いずれの場合もあらかじめCDSに任意の色を付けておきます。

どれぐらい似ているときに似ていると考えるかのしきい値として、相同性(%)とクエリカバレージを使用します。これらはそれぞれ環境設定の画面右上と、generalタブ中で設定することができます。
相同性スコアテーブルを作る
パネルのコンテクストメニューから「creat similarity score table」を実行すると、コンテクストメニューを呼び出したパネルの指定範囲内の全CDSが、全てのパネル(自身も含む)の表示範囲のCDSとblastpで比較した結果を表示します。出力は上から、1)クエリと各クエリが各パネル中の配列と比較した時の相同性(%)、2)相同性を示したCDSの(sbjectの)情報、3)比較の生データ、です。

図は、3つのパネルがあり、そのうち14個のCDSがあるパネルから「creat similarity score table」を実行した結果を表計算シートに貼り付けたもの。
2D viewで総当たり比較する
各パネルの指定範囲が総当たりで比較されます。このとき比較に用いられるプログラムは、プログラム指定プルダウンメニューで選択されているプログラムです。総当たりで比較した後で、各比較イメージのコンテクストメニューからプログラムを選び直して再比較することができます。
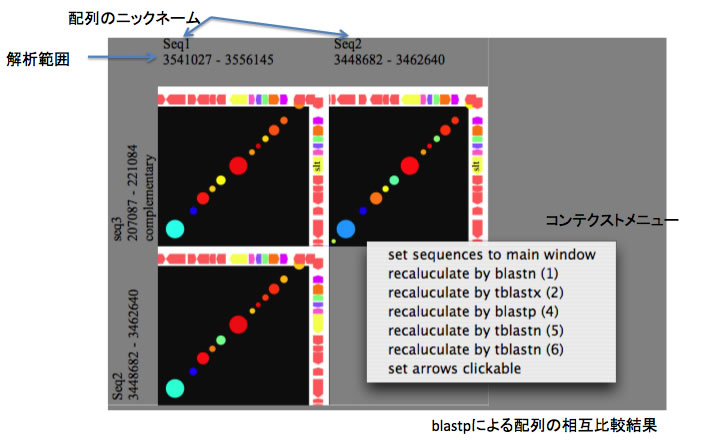
「show symbols in 2D view」スイッチをオンにしておかないとシンボルが表示されません。
「clickable symbols in 2D view」スイッチをオンにしておかないと、シンボルをクリックしても情報が表示されません。これは大量のシンボルを表示することで処理速度が低下してしまうのを防ぐためです。シンボルをクリックできるようにするには、このスイッチをオンにするか、描画イメージのコンテクストメニューから「set arrows clickable」を選択してください。
画面の端に解析範囲と配列のニックネームが表示されています。
コンテクストメニューから、配列と解析範囲をメイン画面に持ち込んで解析することができます(「set sequences to main window」メニュー)。
アノテーションパネルを利用する
アノテーション支援用の機能を利用するには、「add annotation panel」ボタンをクリックしてアノテーションパネルを追加します。アノテーション支援機能についてはこちらを参照してください。
ヒントと注意
解析範囲が長ければそれだけ解析に時間がかかります。実行しようとしている解析がどれぐらい時間がかかるのかを把握しながら解析してください。
blastallやbl2seqのパラメータは環境設定画面で参照/変更できます。
各パネルにカスタムアノテーションを追加できます。パネルのコンテクストメニューから「add / remove annotation」を選択してください。入力形式はメイン画面での形式と同じです(こちら)。
(メイン画面と異なり)カラースケールを変更すると直ちに表示が変更されます。「identity score threshold」およびクエリカバレージについても同様に直ちに変更されます。