ExtractFromGenBankFile
GenBank形式のデータを表計算シートに適合する形に変換する機能です。指定したfeature keyおよびqualifierに関連するデータを抽出します。
はじめに
GenBank形式で記述された注釈は、数件のエントリについて解析するのなら良いのですが、たくさんのエントリについて処理するとなると大変です。例えばGenBank形式のゲノムデータから全アミノ酸配列を抜き出すとなると手作業では無理です。ExtractFromGenBankFileはGenBankファイルを指定すると、その中で使用されている全てのfeature keyと、qualifierを調べその一覧を表示します。ユーザーは抽出したいfeature keyとqualifierをテキストフィールドで編集し、実行ボタンを押します。この簡単な操作によってGenBank形式の中の指定した情報が、表計算シートに適合する形で表示されます。
使用例
「GenBank/DDBJファイルを選択」ボタンを押してGenBankファイルを指定した直後の様子です。このファイルの中で使用されている全てのfeature keyとqualifierが左側のテキストフィールドに出力されています。
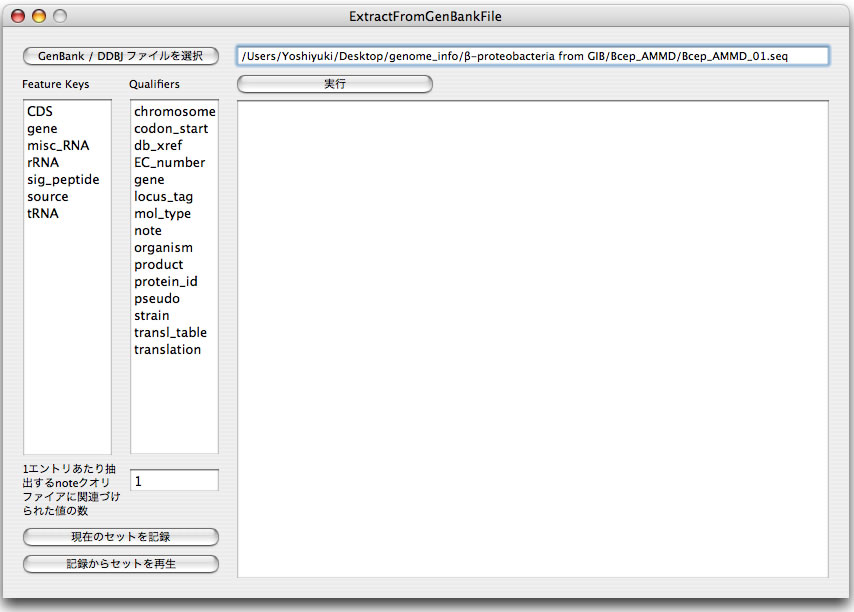
何の情報が欲しいかによって、feature keyとqualifierを編集します。例えば、CDSおよびrRNAおよびtRNAについて、product、locus_tagについて抽出するには下図のように編集します。編集中にスペース文字などの不可視文字が非意図的に入ってしまわないように気をつけてください。
実行ボタンを押して、出力された結果を表計算シートに貼り付けます(下図)。

エントリの登録位置(配列中の位置)も出力されます。GenBankファイル中のエントリの位置情報にcomplementと書かれていた場合は向き-1が、そうでない場合は向き1が出力されます。常にstart_location < end_locationです。左からlocus_tag、productの順に並んでいますが、これはqualifierを編集したときの順番と同じになるようになっています。
注意とヒント
エントリによっては、複数の位置情報を持っています。例えば、エントリの位置情報としてjoin(120..150,180..300,1000,1200)と書かれているような場合です。この場合、これらの数値の中の最小の値と最大の値が位置情報として出力されます。またこの際、一番右側の「CAUTION」列にもともとどのような位置情報が記載されていたのかが表示されます。また位置情報に<や>がある場合(これらはそのエントリの5'あるいは3'の位置が明確でない場合に使用されます)はCAUTION列あるいはその右隣の列に注意情報が表示されます(この際、上の図のB、C、D列にはこれに関する情報は何も表示されません)。
環状レプリコンでは、塩基番号1をまたぐようなエントリはうまく処理されません(例えば20111bpの環状プラスミドのjoin(20000..20111,1,3000)は、B、C、列に、1,20111と表示されてしまいます)。
エントリによっては2つ以上のnote qualifierを持っている場合があります。ファイルを読み込んだときに、最もたくさんのnote qualifierを持っているエントリーのnote qualifierの数が、画面左下に「1エントリ当たり抽出するnoteクオリファイアに関連付けられた値の数」として表示されています。例えば最大3件ある場合では、3と表示されます。このまま実行すると、カラムヘッダーとしてnote_1、note_2、note_3が用意されます。複数件の「note」 qualiferに関してはこのように処理されるのですが、note以外のqualiferについては複数圏ある場合に2件目以降が無視されます(note以外のqualifierが2つ以上あるのを見たことがありません)。
feature keyとqualifierそれぞれのセットは1件だけ保存することができます。「現在のセットを記録」ボタンを押してください。保存したセットを呼び出すには、「記録からセットを再生」ボタンを押してください。
各エントリの位置に対応するDNA配列情報が必要な場合は、「SequenceRetriver」をご利用下さい。