ArcWithColor
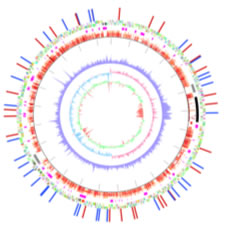
数値データを基に弧など円にそったグラフィックスを作成することができます。サーキュラーゲノムマップ、プラスミドマップの作成に有用です。4種のデータについて(GC-skew、GC content、dinucleotide signature、他配列との相同性検索結果)簡便に描画できるように描画用のデータを作成する機能もついています。このソフトはGenomeMatcherの付属機能ではなく、独立したソフトです。ダウンロードページよりご入手下さい。
はじめに
ArcWithColorはゲノムマップやプラスミドマップのベクターブラフィックスを描画するための機能です。描画できるグラフィック要素は「弧」と「円の中心を通る直線の一部」に限定される反面、表計算シートで編集した大量のデータを入力できる仕様になっており、お手持ちの大量のデータをグラフィックスに変換することができます。また4種のデータについて(GC-skew、GC content、dinucleotide signature、他配列との相同性検索結果)は、描画に必要なデータを自動生成する機能がついています。描画位置(円の中心点からの距離)と線の太さを共有するデータはセットとしてまとめられ、各セットについてのこれらの設定は簡単に変更できます。
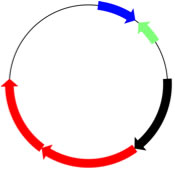
2つのデータタイプ
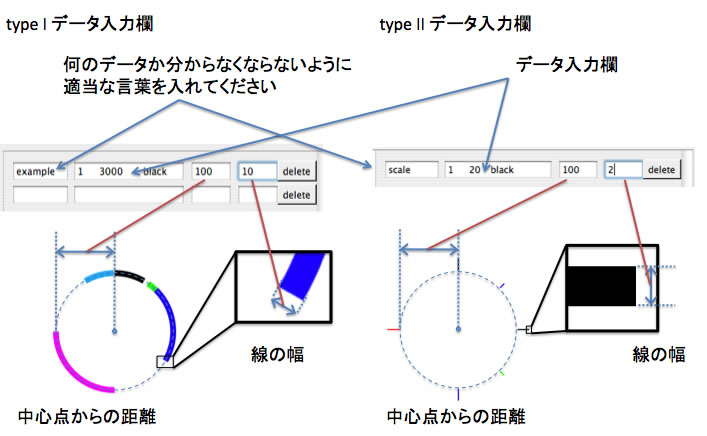
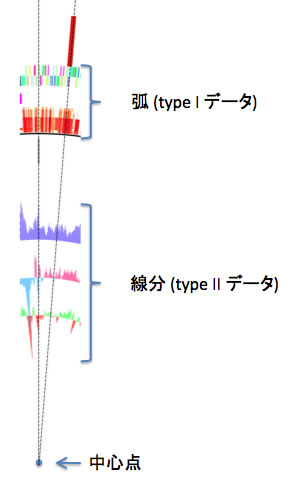 ArcWithColorが描画するグラフィックスには「中心点」が1つあり、全ての描画要素がこの中心点と関連があります。描画できる描画要素は「この点を中心とする円の一部(弧)」と「この点を通る直線の一部(線分)」のどちらかです。前者を描画するのに必要なデータを「type I データ」、後者については「type II データ」と呼びます。右図をご覧いただけると、このグラフィックスの全てがどちらかのタイプの描画要素でできていることがおわかりいただけるかと思います。
ArcWithColorが描画するグラフィックスには「中心点」が1つあり、全ての描画要素がこの中心点と関連があります。描画できる描画要素は「この点を中心とする円の一部(弧)」と「この点を通る直線の一部(線分)」のどちらかです。前者を描画するのに必要なデータを「type I データ」、後者については「type II データ」と呼びます。右図をご覧いただけると、このグラフィックスの全てがどちらかのタイプの描画要素でできていることがおわかりいただけるかと思います。
描画データの準備
描画データは、1)表計算シートで編集する、2) ArcWithColorのデータ生成機能を使う、のいずれかで準備してください。2つのデータタイプごとに入力の形式が異なります。ここでは表計算シートで編集するときについて、2つのデータタイプごとに説明します。どちらのデータタイプについても「中心からの距離」と「線の太さ」を共有するデータをひとまとめに編集してください。「中心からの距離」と「線の太さ」については後で設定します。
type Iデータの編集方法:
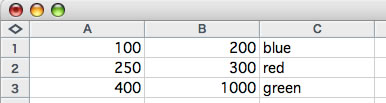
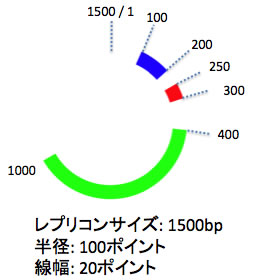
各弧について開始位置、終了位置、線に設定する色のカラーコードを指定します(下図の例では、3つの弧データを編集しました。100塩基から200塩基に「blue」の弧、250塩基から300塩基に「red」の弧、400塩基から1000塩基に「green」の弧を指定しました)。塩基1番は12時の位置に置かれ、配列は右回りです。
type IIデータの編集方法
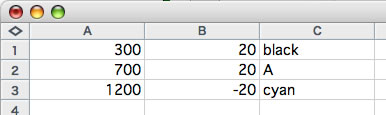
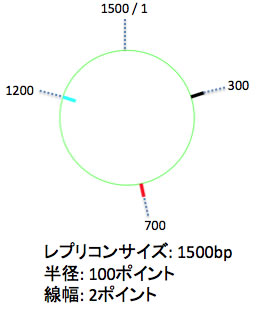
各線分について、描画位置、線の長さ、線に設定する色のカラーコードを指定します(下図の例では、3つの線分データを編集しました。300塩基から20ポイントの長さで「black」の線分、700塩基から20ポイントで「A」の線分、1200塩基から、円の中心に向かって20ポイントの長さの「cyanの線分」を指定しました)。線の長さとしてマイナスの値を指定すると、「中心からの距離」から、中心点に向かって線分が引かれます。プラスの値を指定すると、「中心からの距離」から、中心点から離れる方向に向かって線分が引かれます。
データ生成機能
4種の解析結果を、描画に適した形式で出力する機能がついています。いずれの解析の場合でもまず「main sequence」ボタンから、これから解析するDNA配列(主配列)を含むファイルを指定します。また出力データはそのままデータ入力欄にコピーペーストして入力することができます。
bl2seq (type Iデータを生成)
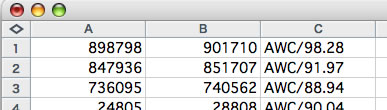
「select targets」ボタンから比較対象のDNA配列を含むファイルを指定し(複数指定可)、「run bl2seq」ボタンを押すと、blastn(bl2seq)で主配列を比較対象のDNA配列と比較したときの結果が出力されます。出力結果を表計算シートに貼り付けた時の例を下に示します。一番上の行は、主配列の898,798塩基目から901,710塩基目までの配列が、比較対象の配列中のどこかと98.28%の相同性を示したことを示しています。相同性スコアの前に「AWC/」がついていますが、数字の前に「AWC/」がつくとその数字はカラースケール中の色を示します(これはArcWithColor特有の設定で、他のGenomeMatcherの機能ではそうではありません)。
![]() カラースケールの例
カラースケールの例
GC-content (type IIデーターを生成)
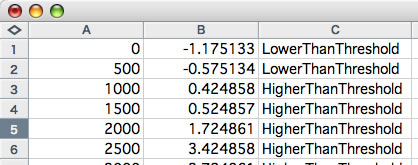
4つの値を指定して(window size、step size、magnification ratio、average)、「run GC-content」ボタンを押します。averageを空欄にすると、配列全体のGC含量が自動的に入力されます。配列の各window中のGC含量についてaverageとの差が表示されます。またaverageより大きいとカラーコードとして「HigherThanThreshold」が、小さいと「LowerThanThreshold」が出力されます。以下にwindow size 1000, step size 500、magnification ratiio 1で実行したときの出力例を示します。1行目は0塩基目の周囲±500塩基(DNA配列の最後500bpと最初500bpのあわせて1000bp)が、averageより1.175133%低かったとのデータです(平均GC含量は65.76%でした)。
GC-skew (type IIデーターを生成)
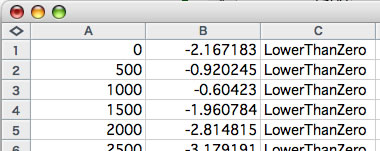
3つの値を指定して(window size、step size、magnification ratio)、「run GC-skew」ボタンを押します。配列の各window中のGC-skew値にmagnification ratioを乗じた値が出力されます。値が0より大きいとカラーコードとして「HigherThanZero」が、小さいと「LowerThanZero」が出力されます。以下にwindow size 1000、step size 500、magnification ratiio 100で実行したときの出力例を示します。1行目は0塩基目の周囲±500 塩基のGC-skew値を100倍した数値が-2.167183であったとのデータです。
dinucleotide bias (type IIデーターを生成)
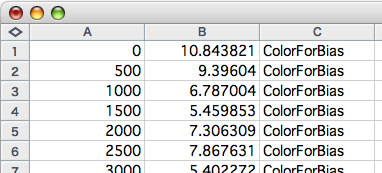
dinucleotide biasについてはこちらを参照してください。3つの値(window size、step size、magnification ratio)と、biasデータとして何を使うかをプルダウンメニューから選択してください。「bias difference from input DNA」を選択した場合は主配列全体のbias dataが計算され、各window中のbias dataとの差(bias difference)にmagnification ratioが乗じられた値が出力されます。「bias difference from user data」を選択した場合は、右のテキストフィールドに入力されたbias dataと各window中のbias dataとの差(bias difference)にmagnification ratioが乗じられた値が出力されます。以下にwindow size 1000, step size 500、magnification ratiio 100で実行したときの出力例を示します。1行目は0塩基目の周囲±500 塩基のdinucleotide bias differenceを100倍した数値が10.843821であったとのデータです。カラーコードとしては常に「ColorForBias」が出力されます。
描画データの入力
type Iデータは画面左側、type IIデータは画面右側に入力してください。入力フィールドが足りない場合は、「add new data (type I)」ボタンあるいは「add new data (type II)」ボタンを押してください。描画データは、上から順番に描画されます。
下の方にあるデータが上の方にあるデータを隠してしまうことがあります。データ入力パネルをドラッグして上下の序列を変更することができます。
各データセットについて「中心点からの距離」と「線の幅」を指定します。これらの値を変更して、絵の見栄えを調節してください。
右側3つのテキストフィールドに入力が完了すると、データの入力欄が消え (処理速度の向上のためです)、右側の2つの背景が緑色になります。
データの入力欄を表示するには、緑色のテキストフィールドのどちらかの値を消して下さい。
描画
主配列の大きさ(bp)と、描画キャンバスのサイズ(ポイント)を指定した上で、「draw」ボタンを押します。グラフィックスをファイルに保存するには「save image to PDF file」ボタンを押します。
ヒントと注意
ここで例として示したのは数件のデータですが、数千~数万件のデータを入力して複雑な絵を描くことができます。
弧が十分に短いときは、実際には円弧ではなく線分として描画されます。
type Iデータについて「開始位置、終了位置、向き、カラーコード」の4つを指定するとarrow headのついた弧が描画できます。向きは、-2、-1、0、1、2のいずれかで指定してください。
現在PDF形式のマニュアルがご利用可能です。ダウンロードページよりダウンロードしてください。
テキストフィールドについてはこちらを参照してください。
blastnの結果を描画することで、類縁ゲノムにない部分が際だちます。
入力したデータは保存/読み込みできます。
ArcWithColorではGenomeMatcherと同様の「色を指定する仕組み」を採用しており、文字と色の関係が定義付けられています。ArcWithColorではカラースケールが1つしかない点、/AWCに続いて数値があると、カラースケールでの色を意味する点、の2点でGenomeMatcherと異なります。