ここではGenomeMatcherとその付属機能について、その作成コンセプトなど各機能に共通する事項について説明します。
色(カラースケール) / 色(カラーコード) / 表計算シート / 入力形式 / 出力形式 / 読める配列、読めない配列 / グラフィックス / 実行上の制約 / MAFFTとMUMmer / アノテーションの入力・出力 / シンボルの表示と非表示 / シンボル / blastpのヒット:どれぐらいのヒットが有意か? / 相同性スコア分布 / 配列Xと配列Y / 翻訳テーブル
GenomeMatcherでは相同性の高低を色調で表現します。色と相同性の関係を表すスケールをカラースケールと呼びます。カラースケールの色調は6つの数値によって決まるようになっています。既定のカラースケールを使うこともできますが、6つの数値を変更してオリジナルのカラースケールを作ることもできます。6つの数値と配合される赤緑青の関係は下図のようになっています。
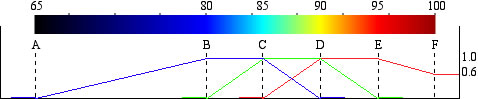
相同性は、2つあるカラースケールのいずれかで表されます。1つはDNAの、もう一つはタンパク質の相同性を示すためのカラースケールです。これはDNAとタンパク質でblastnやblastpなどによって検出される相同性に大きな開きがあり、共通のカラースケールが適用しにくいためです。DNA間の相同性であるか、アミノ酸配列間の相同性であるかによって自動的にどちらかが選択されます。
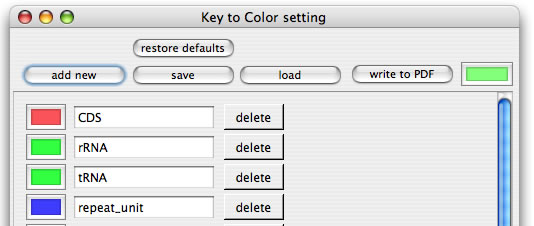
GenomeMatcherでは「色」とその「名前」を定義することで、好きな色を好きな名前で呼び出すことができます。色に付けられた名前のことをカラーコードと呼び、カラーコードは自由に付けることができます。色とその名前の関係は、コマンド+Kで開くwindow中で編集できます。 定義されていない名前を使用すると、デフォルトの色が使われます(下図で右上の色です)。カラーコードは表中でユニークである必要があります。また凡例作成用に、色と名前の関係をベクターグラフィックスで書き出すこともできます(「write to PDF」ボタン)。
GenomeMatcherとその付属機能は表計算シートを併用することを前提にしています。これは表計算シートがデータの処理、保管、共有に極めて適しているためです。ただし表計算シートにも処理しがたいケースがあり、そのようなケースに対処するための機能(RecordMatcherやDataCounter)を付属機能に追加しています。より効率良く解析ができるように、エクセルヒント集のページもご覧ください。
あちらこちらでユーザーのデータを入力ためのテキストフィールドがありますが、多くの場合、指定形式に従って表計算シート上でデータを編集した後に、それを入力欄にペーストしてボタンをクリックすることで入力を完了するようになっています。指定形式は守っていただかなくてはなりません。また、意図せずに関係のないテキストがテキストフィールドに入力されていないように気を付けていただく必要があります。このような仕様になっていることで、大量のデータを一括して入力することができます。指定形式は、その入力テキストフィールドのそばに書いてあるか、周辺にツールチップ(ヒント)が埋め込まれていますのでご参照下さい。
また各種プログラムのパラメーター設定では、基本的に各パラメーターを一つのスペースで区切ってください。
出力データの見た目が整列されていない場合があります。これは出力データを表計算シートにペーストして利用することが前提になっているためです。出力データをセレクトオール後、コピーして、表計算シートに貼り付けてください。
GenomeMatcherが受け付けるファイル形式は 1) DDBJ / GenBank形式 (ただし複数の異なるDNA配列が含まれているモノを除く)、 2)FASTA 形式、3)テキスト形式のDNA配列、です。機能によってはmulti FASTA形式を読み込めますが、多くの場合読み込めません。その他の形式は読みこめません。読み込めるはずの形式が読み込めないときはこちらをどうぞ。
基本的にベクターグラフィックス(PDF形式)で出力できるようになっています。メイン画面の画像は、「イメージ」メニューから選択してください。それ以外の場合は各機能のウィンドウにあるPDFと書かれたボタンをクリックしてください。
GenoemMatcherは、長時間かかる解析や多量のメモリを食いつぶしてクラッシュすることが予想されるような解析を始めてしまうおそれがある場合は解析実行ボタンをグレイアウトさせる仕様になっています。環境設定のlimitタブでどのような制限が設けられているか確認/変更することができます。設定を変更する際には十分にこの点をご理解ください。
 各種の制限値
各種の制限値
またMAFFTプログラムとMUMmerプログラムについてはこちらもどうぞ。
GenomeMatcherで MAFFTおよびMUMmerを実行するにはユーザー自身の手でこれらのプログラムをお使いのMacにインストールする必要があります。またインストールしたこれらのプログラムのインストールした場所を、環境設定のMUMmerおよびMAFFTタブ中で指定する必要があります。
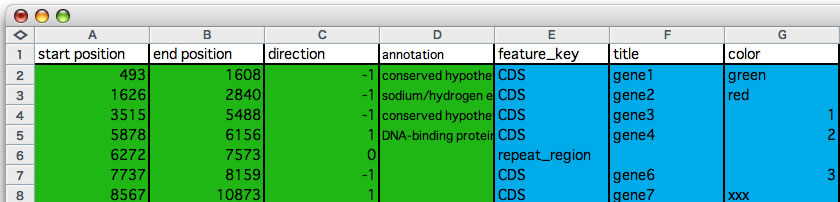
GenomeMatcherでは、ユーザーのカスタムアノテーションを入力して表示することができます。アノテーションを入力するにはアノテーション入力の際の指定形式について理解して頂く必要があります。指定形式は、コマンド+Mで開くwindow中で確認することができます。入力形式についてはこちらも参照して下さい。下図にアノテーションの編集例を示します。注意点は以下の通りです。
- start positionはend positionよりも常に小さい数字になるようにして下さい。
- directionは1(正向き)、-1(逆向き)、0(向き属性なし)のいずれかで指定して下さい。
- feature_keyには表示、非表示の設定が適用されますので気をつけて下さい。表示する指定になっていないfeature_keyを指定すると、その注釈は表示されません。feature_keyを省略すると「*」で代用されます。
- タイトルはシンボルに表示される文字です。表示スペースに余裕があるときのみ表示されます。
- 最初の2列のみが必須です。
- 表計算シート中、タイトル行は含めないで下さい。
- メイン機能と付属機能「compare sequences」で、入力形式は同一です。
アノテーションを出力するには、メイン画面では「解析」メニューの中の「選択範囲のアノテーションを見る」から、「CompareSequences」ではコンテクストメニューの「see anotation in selected range」を選択します。いずれの場合も表計算シートに適合する形式あるいはGenBank形式で見ることができます(カスタムアノテーションはGenBank形式になりません)。
DNA配列には色々な注釈が付いていると思いますが、それぞれの注釈には1つのfeature keyが関連づけられています。GenomeMathcerでは指定したfeature keyを持つ注釈だけを表示する設定になっています。なぜかというと、例えばfeature keyの一つに「source」があり、これは配列位置1から最後までを位置情報として持っていますがこのようなfeature keyが表示されると見にくくて仕方がないからです。feature keyに関する表示、非表示の設定は環境設定のsymbolsタブで変更できます。
また、注釈情報を表すシンボルは、表示範囲がある程度短い時のみ表示されるようになっています。これはたくさんのシンボルが表示されるようになると動作が遅くなること、シンボルがあまり大きく表示されないような場合では、表示する意味があまりないことが多いことによります。表示範囲の長さと、どのfeature keyを表示するかについては2通りの設定ができます(環境設定のsymbolsタブ中)。
注釈情報の存在は、矢印あるいは長方形などのシンボルで表示されます。シンボルをクリックすることで注釈を参照できます。また、コマンドを押しながらクリックすると、シンボルの色、タイトル、向きを変更するためのwindowが開きます(下図)。またコンテクストメニューもあります。 シンボルのタイトルはスペースに十分余裕があるときのみ表示される設定になっています。なお、GenBank形式のファイルを読み込んだとき、遺伝子名があれば遺伝子名が、なければlocus tagがタイトルとして表示されます。
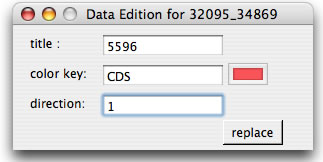
シンボルの例:タイトルは5596。feature keyは「CDS」。変更を反映するには「replace」ボタンを押します。
GenomeMatcherではblastpをかけたときにクエリカバレージ(環境設定のgeneralタブ中で設定)をしきい値にして、ヒットと見なすか否かを決めています。また、環境設定の右上にあるテキストボックスで、相同性のしきい値をセットすることで相同性が低いヒットを非表示にすることができます。
解析を始めてから終了するまでの間に得られたヒットのスコア分布図が環境設定で表示されます。クエリカバレージを設定してblastpを実行した場合は、クエリカバレージをクリアしたヒットの分布図になります。分布図のコンテクストメニューから、分布図をPDF保存したり、分布図を作成する上での生データを取得することができます。
 相同性分布図とそのコンテクストメニュー(生データにアクセスできる)
相同性分布図とそのコンテクストメニュー(生データにアクセスできる)
メイン画面で「select file(X)」のボタンを押して読み込んだDNA配列のことを配列X、「select file(Y)」のボタンを押して読み込んだDNA配列のことを配列Yと呼びます。
環境設定の「general」タブ中のプルダウンメニューから翻訳テーブルを選択してください。