研究経過
【原著論文】少数派の利益に関わる変異を探すためのGWAS(清水班)
April 6, 2023 2:18 PM
Category:論文発表
main:清水班
計画研究・清水健太郎教授グループ(チューリッヒ大学・横浜市立大学)の佐藤安弘上級助手らが千葉大学との共同研究で行った頻度依存選択のゲノムワイド関連解析(GWAS)が米国進化生物学会誌Evolutionに発表されました。
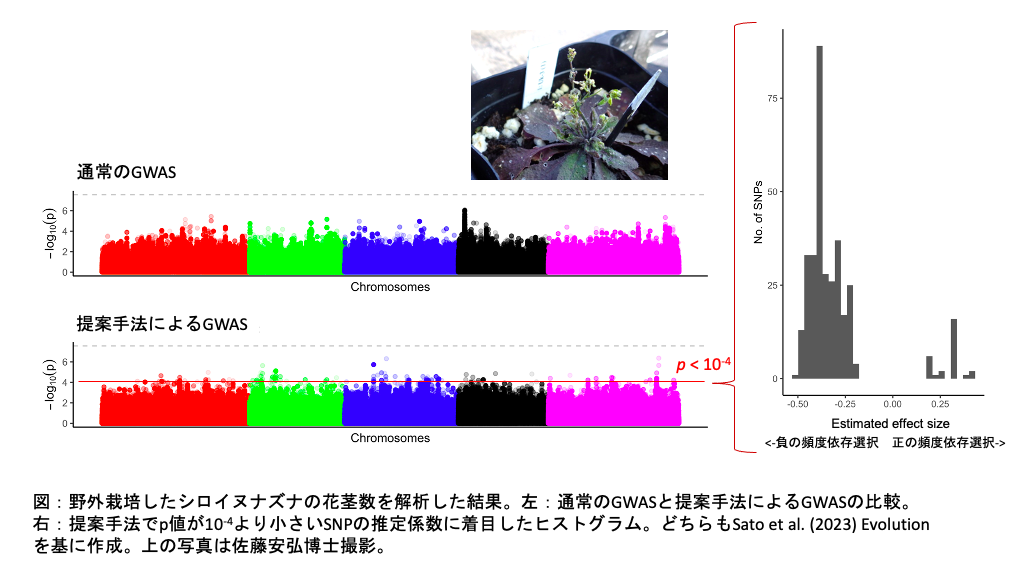
頻度依存選択とは特殊な自然選択圧の1つで、少数派の対立遺伝子が有利になる負の頻度依存選択と、多数派の対立遺伝子が有利になる正の頻度依存選択の2つがあります。特に負の頻度依存選択は、少数派の変異が集団から排除されるのを防ぐので、遺伝的多様性を安定的に維持する大事な要因です。例えば、病原抵抗性の進化では、新たに生じた少数派の変異が病気から逃れるために有利になることがあります。植物の生殖に関わる形質でも、送粉者を介した負の頻度依存選択によって異なる花の色のランが共存できることが知られています。
これまでに我々は、統計力学のイジングモデルを用いて、個体間相互作用を取り入れたGWAS手法NeighborGWASを開発してきました(Sato et al. Heredity 126:597-614, 2021, https://www.nature.com/articles/s41437-020-00401-w) 。今回さらに、 1遺伝子座-2対立遺伝子座からなる頻度依存選択のモデルに拡張しました。そして、野外栽培したシロイヌナズナの花茎の数を解析しました。その結果、花茎数と相関が強かったSNPには正よりも負の頻度依存選択に関わるものが多く見られました。さらに、これらのSNPの近くには病原菌抵抗性に関わる遺伝子が見られたことから、負の頻度依存選択に関わる既知の機能と比べても妥当な結果が得られたと考えられます。
ここまで読んで、植物の生殖に詳しい方々は「自家不和合性システムに対する負の頻度依存選択にも使えないのか」と問われるかもしれませんが、現状では2対立遺伝子までしか対応できていません。3型以上の場合にもSNPマーカーを基にしたGWASで検出できるかは今後の課題です。
<発表論文>
Yasuhiro Sato, Yuma Takahashi, Chongmeng Xu, Kentaro K. Shimizu,
Detecting frequency-dependent selection through the effects of genotype similarity on fitness components.
Evolution, Volume 77, 1145-1157, 2023