設定パラメーターの詳細
ここでは、「general settings」ボタンを押して開くウィンドウ中の各タブで設定できるパラメーターについて説明します。

「folder」タブ
解析に使われるファイルの名前はデフォルトでは454ContigGraph.txt、454AllContigs.fna、454AllContigs.qual、454Scaffolds.txt、454Scaffolds.fna、454Scaffolds.qual、454PairStatus.txt、454TrimmedReads.fnaです。必要があればファイル名を変更できますが、必要ないでしょう。
「contigs」タブ
コンティグの中にはカバレージが低くく、「ゴミ」であると考えられるものがあります。またコンティグとコンティグをつなぐリードが少ない繋がりは意味のない繋がりである可能性があります。このタブ内では、カバレージおよびリード数に基づいて、コンティグあるいは「繋がり」を無視するかしないかのしきい値を設定することができます。設定変更は「run」ボタンをクリックする前のみ有効です。
意味のないコンティグや繋がりを排除しないでおくと、メモリを多大に消費するだけでなくコンティグ間の繋がりなどを理解するのが困難になることがあります。一度「run」し、その結果を見てこの値を設定し直して再度runするようにしてください(runした後に変更しても、変更は無効です)。
「appearance」タブ
コンティグを結ぶ線の太さについて設定できます。0.1としておけば、コンティグをつなぐリード数が100の時に10ポイントの線幅の線が引かれます(100 X 0.1 = 10)。またイルミナのペアデータがある場合は、ペアを結ぶ灰色の曲線が表示されます。その線の太さを同様に指定できます。
「edge/node」タブ
コンティググラフを自動で作成する際、各コンティグは、「edge」あるいは「node」に振り分けられます。その際、4つの判断基準が用いられます。4つの基準には優先度があり、優先順に、
- 1: どちらかの末端が孤独末端であるものはedgeである。
- 2: 「指定した長さ」以下のコンティグはnodeである。
- 3: 「指定した長さ」以上のコンティグはedgeである。
- 4: 両末端とも繋がり先が1つのコンティグしかないものはedgeである。
- 5: 以上できまらない場合はnodeである。
となっています。
各コンティググラフ中ではコンティグからコンティグへのルートが探索されます。この際何ステップ先まで探すかの設定があります。この設定はContigGraph1およびContigGraph2の両方で有効です。これはループ箇所で無限ループに入るのを防ぐための設定です。なお一度作成されたコンティググラフであっても、この設定を変更した後「find route again」ボタンを押すと、同コンティググラフ内にあるコンティグについて、ルートの再探索が実施されます。その時点でコンティググラフ内にないコンティグに関してはこの際のルート再探索の対象となりませんので気を付けてください。
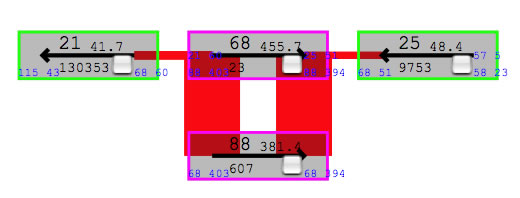
無限ループになりうる箇所の例。21-68-(88-68)n-25。
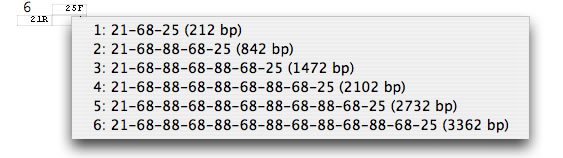
なお上の例ではサザン解析によりn = 10であることが明らかになったが、デフォルトの「12」ではn = 10に対応するルートが表示されない。このような場合に、「12」を設定し直して「find route again」ボタンを押すと、十分な経路が探索される。闇雲にこの数値を大きくすると、計算時間の増大などの不都合が生じるので注意が必要。
「PCR product size」タブ
454Finisherは、primer総合情報シートを出力します。総合情報シートの中には、各primerをどの96-well プレートのどの番地に入れるかについてのオススメが含まれます。なお予想プロダクトサイズがあまりにも異なるPCR反応を同じ96-wellプレートで実行するのはあまり賢い方法ではありません。そこで454Finisherが出力するオススメでは、予想されるプロダクトサイズに基づいてついて3通りのプレートに、分注先を指定するようになっています。このタブ内では予想PCRプロダクトサイズに基づいて、どのPCRプレートに分注するかを決定するための値を2つ指定できます。1200と3000を指定すれば、1200以下のものは「plate short」に、1200から3000のものは「plate medium」に、3000以上のものは「plate large」に割り振られます。
なお1つの課題シートに含まれるPCR反応は、1つの種類のプレート上で実行するように指定されます。どのプレートに分注するかについては、各課題シート中のPCR産物のうち最大のプロダクトサイズに基づいて決定されます。
「blastall」タブ
各コンティグを全コンティグに対してblastnで比較するときに用いるオプションを設定します。特にこだわらなければ何も指定しないでください。
カラースケールは、blastnの結果をどのような色で表示するかの指定です。これも特に変更は必要ないように思います。
「scaffold」タブ
スキャフォールドが既に得られている場合、454Scaffolds.txtに記述されているデータに従って、特定のコンティグから特定のコンティグへのルートを探索します。例えばcontig00029からcontig00030をつなぐルートを検索する場合、これらコンティグをつなぎうる全てのルートが探索されます。このとき、ある限度を超える長さのルートを無視するようになっています。ここで指定するのはこの限度の値です。なおこのルート探索の際、「edge/node」タブで設定される何ステップ先まで探すかの値も有効です。
「alert」タブ
各種アラートを出力するかどうかの設定です。
「pair read」タブ
「external (non-454)pairs」ボックスでは、454以外(主にイルミナリード)のペアリードの取り扱い方について設定します。「run」ボタンを押した際に、(1)探索するかどうかの設定、(2)エッジ(重複コンティグでないと推測されたコンティグ)についてのみ探索するかどうかの設定、また、コ(3)ンティグの末端何塩基の範囲を検索するかについての設定をします。このときの探索では100%一致するもののみを探します。また、100%一致するようなものが他のコンティグの探索範囲にも存在する場合、そのようなリードにそのものは出力するようになっていますが、そのペアについては出力されないようになっています。(3)については、ペアの間の距離をもとに設定してください。
「454 pairs」ボックスでは、コンティグ末端について454のリードを探す範囲を指定します。insert 8 kbのライブラリを作成した場合は、8 kbより少し大きい値を指定すると良いでしょう。なお、この際の探索では、454PairStatus.txtファイルの記述に基づいてコンティグの末端に位置するリードを得ています。
「external programs」タブ
GenoFinisherは外部プログラムとして、blastall、conserv、velvet、abyssを使用しています。このうちvelvetとabyssについてはユーザー自身で実行ファイルを入手していただく必要があります。velvetについては、「velveth」と「velvetg」を、abyssについては「ABYSS」をご入手ください。velvethとvelvetgは同じフォルダ内に置いた上で、そのフォルダへのパスを指定してください。ABYSSについては、ABYSSへのパスを指定してください。
これらの実行ファイルが手に入らない場合、ソースファイルを入手後、それをコンパイルする必要があります。コンパイルをするにはコンパイラが必要ですので、まずコンパイラをインストールしてください。Macを購入したときに付属したDVDに入っている「Xcode」をインストールすると、コンパイラがインストールされます。この方法が簡単です。実行ファイルの入手/ソースファイルのコンパイルが困難な場合はご相談ください。
その他
ペアと認識されるリードID
2つのリードがペアであるかどうかは、一方のリードIDについてペアとなるIDを求めて、もう一方のリードIDと等しいかどうかで判別しています。ペアとなるIDは以下のように求めています。
- 末尾が1で終わるリードIDのペアIDは、末尾を2に変換したもの。同様に、2の場合は1に変換したもの。
- 末尾がrに対してf。fに対してr。
- 末尾が_rightに対して_left。_leftに対して_right。