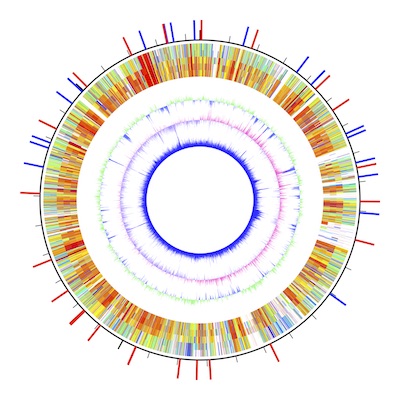

AWCは、数値データを元にサーキュラーマップを描画するためのアプリです。
App Storeからの配信を開始しました(vresion 1.61)。
古いバージョン(ArcWithColor ver.1.5)はここからダウンロードできます。
サーキュラーマップを、数値データに基づいて描画できます(ユーザーがユーザー自身で考えたアルゴリズムで算出した数値データを、グラフィックスに変換できます)。
それ以外にも、AWCにはデータ生成機能がついており、AWCで描画するための数値データを計算することもできます。計算できるのは、
です。
出力されるのはベクターグラフィックス(PDF形式)です。サイズを指定して描画できますので、例えばA0サイズに描画することもできます。大きく描画できるので、論文などで見る「細部まで見ることはないゲノムマップ」とは異なって、実際にデータを詳細に見るのにも使えます。
マニュアルはここからどうぞ。
なるべく解像度を高くしてご覧ください。動画中で使用しているデータはここから入手できます。
説明動画です(5分)。忙しい人用。
説明動画です(15分)
AWCのデータ生成機能を使ってデータを得るか、エクセルシートなどでデータを整形して、これをデータ入力欄に入力してDrawボタンを押します。ベクターグラフィックス(PDF形式)が出力できます。
GenomeMatcherにGeneDrawerというアクセサリーツールがあります。
こちらでは、サーキュラーマップではない「ゲノム的」な絵が、数値データに基づいて描画できます。NGSのリードのマッピング結果とか、in houseの解析とか、です。
AWCで処理可能なものに限らず、データ処理が困難な場合、PIの方よりメールにてご相談ください。特に既存のツールなどで処理できない新しいアイデアに基づくデータ処理のコーディングに興味があります。科研費などプロジェクト分担者などへの参加依頼も歓迎します。
本ソフトウエアは無保証です。バグを見つけましたらご報告いただけますと助かります。
作者: 東北大学大学院生命科学研究科 准教授 大坪嘉行
yoshiyuki.ohtsubo.a6[アット]tohoku.ac.jp
本ソフトウエアを利用した成果を公表する場合、以下の論文を引用してください (AWCはGenomeMatcherプロジェクトに属しています)。
Y. Ohtsubo, W. Ikeda-Ohtsubo, Y. Nagata and M. Tsuda: GenomeMatcher: a graphical user interface for DNA sequence comparison. BMC Bioinformatics 9: 376 (2008)
作者は現在、新型コロナウィルスを2分程度で検出可能な迅速検出法の構築を目指しています。これまでに新型コロナウィルスに結合するDNAアプタマーを取得し、これを複数装荷した分子を作製しているところです。このような分子はコロナルウィルスが存在すると凝集体を形成させ、これを容易に検出できるものと期待しています。これまで複数の研究助成などに応募してきましたが、残念ながら研究費の獲得に至っておりません。この研究を続けるため、私の研究室へのご寄附をご検討いただければ幸いです。
寄附金は、新型コロナウィルス関連の研究ならびにソフトウエア開発などの研究教育を目的とした活動に有効に利用させていただきます(大学によって通常の研究費と同等に管理されます)。寄附金についての本学での取り扱い、税制上の優遇措置などについてはこちらをご覧ください。
ご寄附頂ける場合は、専用の様式にご記入いただき、東北大学生命科学研究科会計係までメールにてお送りください。ご検討のほど、よろしくお願いいたします。
様式はこちらからダウンロードしてください。メールアドレスはlif-kaik[at]grp.tohoku.ac.jpです。
 大学改革の方針転換を。「REUP提案」をお読みください。
大学改革の方針転換を。「REUP提案」をお読みください。
 入試や就活で「推し研」を問いませんか?
入試や就活で「推し研」を問いませんか?
 本書では、科学的素養を7項目で整理・明文化しています。教育の質、学びの質を高め、研究力向上を。
本書では、科学的素養を7項目で整理・明文化しています。教育の質、学びの質を高め、研究力向上を。
 本書では、研究力低迷問題を分析し、解決策を提案しています。研究、教育に携わる多くの方にお読みいただけますと幸いです。解説資料平易版 ・提案資料
本書では、研究力低迷問題を分析し、解決策を提案しています。研究、教育に携わる多くの方にお読みいただけますと幸いです。解説資料平易版 ・提案資料
 QRコードをスマホで読み取って出席登録。出席管理WEBシステムです。
QRコードをスマホで読み取って出席登録。出席管理WEBシステムです。
 TraceViewer。変性ポリアクリルアミドゲル電気泳動の代わりにシーケンサーでデータを取りませんか?macOSアプリです。
TraceViewer。変性ポリアクリルアミドゲル電気泳動の代わりにシーケンサーでデータを取りませんか?macOSアプリです。
 ShortReadManager NGSデータの処理に便利なmacOSアプリです。変なアイコンですみません。
ShortReadManager NGSデータの処理に便利なmacOSアプリです。変なアイコンですみません。
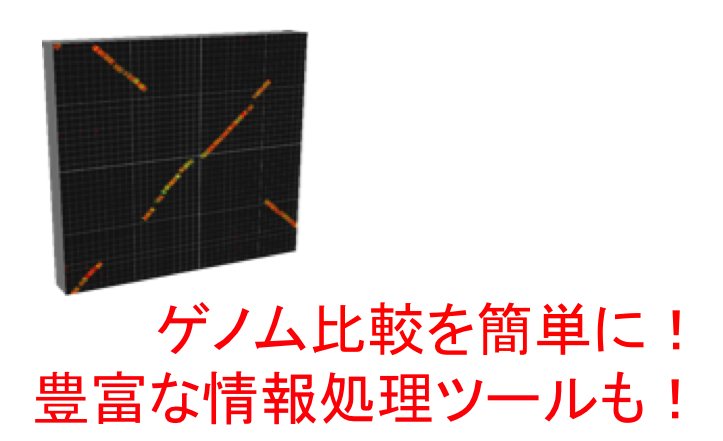 GenomeMatcher ゲノム比較機能をはじめ色々な情報処理ツールがついています。数値データをグラフィックスに変換する機能も。
GenomeMatcher ゲノム比較機能をはじめ色々な情報処理ツールがついています。数値データをグラフィックスに変換する機能も。
 GenoFinisher バクテリアゲノムをshort readだけでも決定可能です。バクテリアゲノムのフィニッシングでお困りの方はご相談ください。
GenoFinisher バクテリアゲノムをshort readだけでも決定可能です。バクテリアゲノムのフィニッシングでお困りの方はご相談ください。
 DNA配列の相補配列を計算する関数を含むエクセルシートです。
DNA配列の相補配列を計算する関数を含むエクセルシートです。
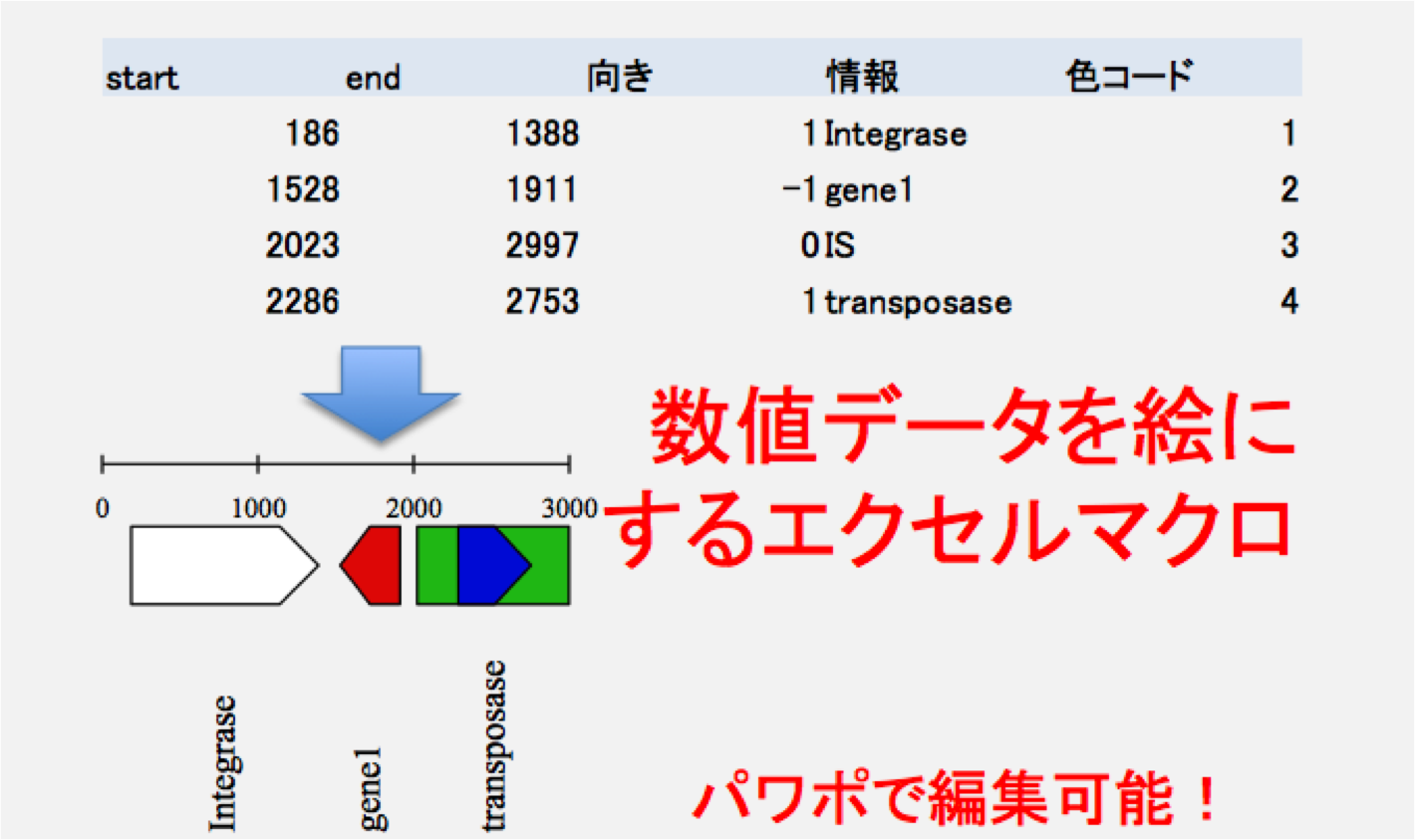 数値データを絵に変換するマクロを含むエクセルシートです。
数値データを絵に変換するマクロを含むエクセルシートです。
 数値データを元にサーキュラーマップが描けます。データ生成機能もあります。
数値データを元にサーキュラーマップが描けます。データ生成機能もあります。
 大学等研究機関での実験機器類の共用を促進するためのウエブシステムです。
大学等研究機関での実験機器類の共用を促進するためのウエブシステムです。
 DNA配列/アミノ酸配列を2次元パネルの上で動かせるソフトウエアです。配列比較も。
DNA配列/アミノ酸配列を2次元パネルの上で動かせるソフトウエアです。配列比較も。
 例の処理を簡単に済ませるあのツールです。
例の処理を簡単に済ませるあのツールです。
 iPhoneアプリです。勤務地への入域と出域時刻のログを自動的にとります。App Storeから入手できます。
iPhoneアプリです。勤務地への入域と出域時刻のログを自動的にとります。App Storeから入手できます。
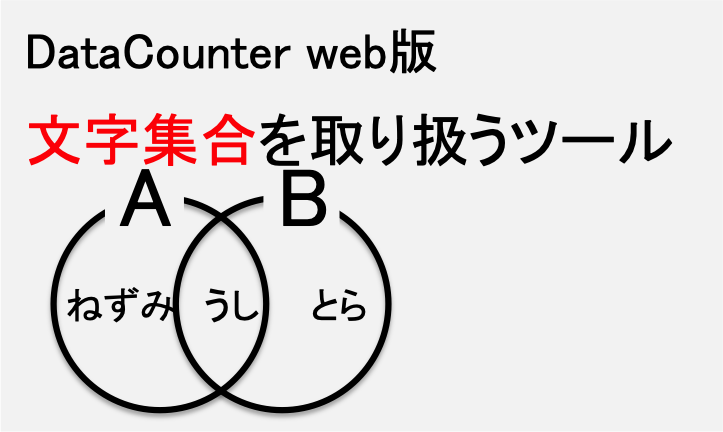 文字列集合を取り扱える便利ツールです。
文字列集合を取り扱える便利ツールです。
 GenBankファイルのデータを、エクセルシートで取り扱えるように変換するツールです。
GenBankファイルのデータを、エクセルシートで取り扱えるように変換するツールです。
 文字列を取り扱える便利ツールです。
文字列を取り扱える便利ツールです。
 作者研究室ホームページ。共同研究の提案と研究室への寄付を歓迎します。
作者研究室ホームページ。共同研究の提案と研究室への寄付を歓迎します。
作者プロフィール: 環境細菌の研究を進める一方で、様々なソフトウエアを作成、公開している。