

SeqViewは、DNA配列を二次元view上で取り扱えるソフトウエアです。App Storeより配信中です。
DNA配列がいつくかあって、お互いにどこが似ているか調べたい時はよくあると思います。DNA配列を二次元viewで動かせたら便利なのではないか?というのがSeqViewの開発動機です。
DNA配列をお互いに比較して、どこが同じであるか表示することができます。相同性の高低は色で表されます。不一致箇所は黒い帯で示されます。
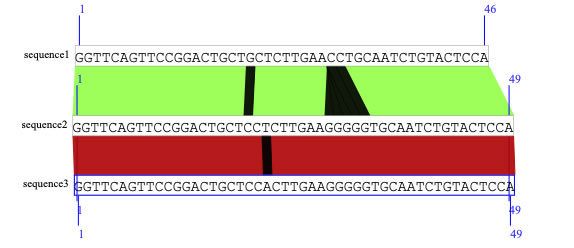
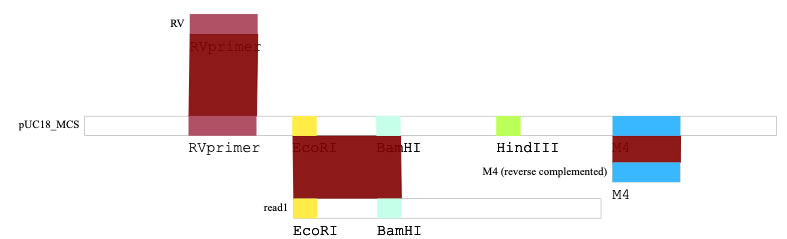
配列に色をつけることもできます。下の図はpUC18のクローニングサイト周りです。

DNA配列はBarとしても表示できます。ゲノム配列など、ある程度長い配列はBarでの表示が良いと思います。
配列の入力方法
左上のテキストフィールド(adding sequencesボックス内)に、マルチFASTA形式のデータをペーストしてaddボタンを押してください。入力しようとする配列はユニークなID(名前)を持つようにしてください。
配列の選択
クリックすると配列が選択されます。選択された配列には青枠がつきます。シフトを押しながらクリックすると、複数選択できます。Viewを左から右にドラッグすると、ドラッグエリアに完全に含まれる配列が選択されます。右から左にドラッグすると、ドラッグエリアに少しでも含まれる配列が選択されます。
コマンドを押しながらクリックすると、「特殊選択」されて、赤枠が表示されます(特殊選択については後述します)。特殊選択されたものをコマンドを押しながらクリックすると特殊選択が解除されます。
配列の移動
ドラッグで動かせます。複数選択された状態で1つをドラッグすると、選択されたものが同じように動きます。
配列のリバースコンプリメント
配列のコンテクストメニューから「invert panel」を選択すると、DNA配列の場合リバースコンプリメントした配列が表示されます(元の配列がACGTTTTTであればAAAAACGTが表示されます)。アミノ酸配列の場合は、逆順に表示されます。
配列の消去
デリートボタンで選択した配列を消します。
blastによる比較
blast+ による比較ができます。配列が複数選ばれた状態で、blast comparisonボックス内にあるrunボタンを押してください。
プログラムセレクタからblastプログラム(blastn、blastx、blastp)を選択してください。 blastパラメーターはよくわからなければ空欄でお使いくはださい。
内部でどのように処理されているかを理解すると、操作がやりやすくなると思います。特殊選択されている配列がない場合は、全ての配列がブラストデータベース化され、全ての配列をクエリとしてblast比較が実施されます。特殊選択されている配列がある場合は、特殊選択された配列がブラストデータベースとなり、選択されたものがクエリとなってblast比較が実施されます。一つ一つのクエリについて、自身に対するヒットを無視(特殊選択されている配列がない場合)した上で、hits to storeで設定した数を超えない数のHSPのうち、指定の3条件(query coverage、length、およびidentity)を満たすものが記録されます。値として0を指定すると、その条件は常に満たすことになります。特殊選択された配列がある場合は、各クエリとなった配列がトップヒット部分に移動します。
Store mismath data チェックボックスにチェックを入れておくと、blast実行時に、HSP中のどこにミスマッチがあったかが保持され、Draw mismathにチェックボックスにチェックが入っているとこのミスマッチが描画されます。
ミスマッチデータはヒット描画部分のコンテクストメニュー「copy mismatch data to clipboard」から取り出せます(下図)。この時、1つ1つのミスマッチ塩基ごとに、アラインメントデータが出力されますので注意してください。

ヒットデータの消去
コンテクストメニューのremove hit..から、消去したいヒットデータを選ぶとヒットデータを消すことができます。また配列を選択した状態で、editメニューから「remove hit data from selected sequences」を選択すると、選択状態の配列に関するヒットデータを全て消すことができます。

配列に色をつける
配列を検索し、見つかった配列に色をつけることができます。find and markボックス中で、検索する配列と、見つかった配列につける色をカラーキーで指定し、find and markボタンを押してください。
例えば、EcoRI GAATTCを指定すると、GAATTCの部分がカラーキー「EcoRI」に関連づけられた色で表示されます。未定義のカラーキーを使用すると、ランダムに色が1つ生成されてカラーキーと関連づけられます。必要に応じて色とカラーキーの関係はコマンド+Kで開くwindow中で編集してください。
両側のストランドを検索するか、指定することができます。またカラーキーを表示するかどうか指定することができます。
findする配列は同時に複数を指定できます。改行で区切って指定したください。
配列の一部分の選択
配列をダブルクリックすると配列の一部分を選択できるようになります。この場合、その部分の位置、選択している長さ、その部分のTm値が表示されます。
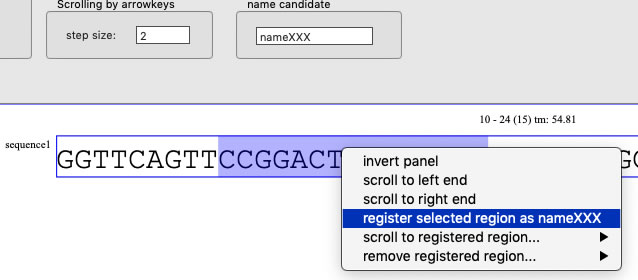
配列の一部分に名前をつけて登録する
配列の一部分に名前をつけることができます。name candidate boxに、名前候補を入力し、配列の一部分を選択した状態で、右クリックから「register selected region as XXXX」を選択します(XXXXの部分はname candidate boxで指定した名前が入ります。名前はカラーキーとしても使用されます。必要に応じてカラーキーに対応する色を変更してください。)。名前はユニークである必要があります(各配列ごとに、その名前は1回しか使えません)。名前をつけた部分には、その配列のコンテクストメニュー「scroll to registered region....」でスクロールすることができます。登録は、右クリックから消去することができます。
スクロール幅の設定
左右の矢印キーで画面をスクロールさせることができます。スクロール幅は、base単位で指定してください。100を指定すると、左右矢印キーをおすごとに100塩基分スクロールします。
配列を探す
View上にある配列はプルダウンメニューから探すことができます。配列がある部分にスクロール後、配列が2回点滅します。
配列の整列
左揃え、右揃え、縦方向に整列ができます。
PDFファイルへの書き出し
FileメニューのSave PDF imageを選択してください。
複数viewの管理
viewを複数保持することができます。各viewにはユニークな名前がつけられている必要があります。viewを追加するにはユニークな名前を指定して、add viewボタンを押してください。
メッセージの表示
画面上にエラーメッセージなど各種メッセージが表示されますので、参照してください 。
Q1: blastの結果が表示されません。
A1: blast comparison枠内の設定は、blast解析実行時の設定です。blastのパラメータが適切か確認してください。わからなければパラメータ欄は空欄にしてください。さらに、appearance枠内のdata sourceセレクタで、どのblastプログラムの結果を表示するか選んでください。
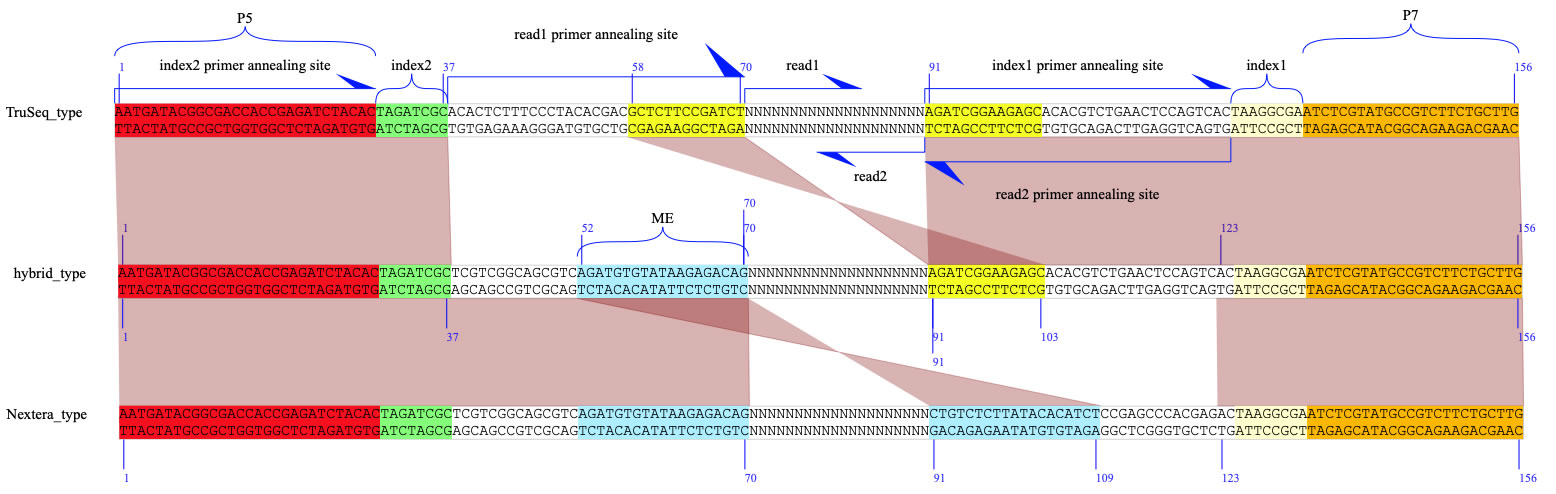
TruSeq型とNextera型があります。PDFはこちら。SeqViewデータはこちら。配列は、イルミナ社の資料から推測したものです。間違い等ありましたらお知らせいただけますと幸いです。
英語版チュートリアルがありますのでご覧ください。App Storeでの審査の都合上、英語です,,,,,,
SeqViewはフニッシング用ソフトウエア「GenoFinisher」および「AceFileViewer」の付属ツールとして作成したものを、フィニッシングから切り離して機能を追加、独立させたものです。フィニッシングでは、複雑なリピート構造の詳細を調べたり、gap前後のコンティグの配列がオーバーラップしている場合に、類縁ゲノムのアミノ酸配列データを使ってその部分がオーバーラップしているかを推測するなどの用途に用いていました。「SeqView」利用におかれましては、「GenoFinisher」および「AceFileViewer」を初めて使用して全ゲノム配列決定をした以下の論文を引用していただけますと幸いです。
Ohtsubo Y, Maruyama F, Mitsui H, Nagata Y, Tsuda M. Complete genome sequence of Acidovorax sp. strain KKS102, a polychlorinated-biphenyl degrader. J Bacteriol. 2012 Dec;194(24):6970-1. doi: 10.1128/JB.01848-12. PubMed PMID: 23209225; PubMed Central PMCID: PMC3510582.vesrion 0.953
描画レートの不具合を解消しました。
日本語マニュアルのページへのリンクを追加しました(Helpメニュー)
(1) 色の設定が保存されない問題を解消。
(2) viewごとに"find and mark"設定ができるようになりました。
(3) registerした領域の名前の表示非表示を選択できるようにしました。
vesrion 0.97 20210705
(1) DNAの二本鎖表示ができるようになりました。ストランドごとに、DNA配列の部分に名前を設定できます。
(2) 入力した配列に対して、これをそのまま表示するか、reverseした配列を表示するか、complementした配列を表示するか、reverse-complementした配列を表示するか、を選ぶようにしました。
vesrion 0.98 20210815 公開
(1) 配列にアノテーションを付することができるようになりました。テキスト + カッコまたは左右矢印です。
(2) 両方のストランドを表示できるようにしました。どちらも選択できます。
(3) 色設定がファイルに保存されるようになりました。
本ソフトウエアは無保証です。バグを見つけましたらお知らせいただけますと助かります。バグをお知らせいただけますと、ユーザーのみなさんが助かります。
作者: 東北大学大学院生命科学研究科 准教授 大坪嘉行
yoshiyuki.ohtsubo.a6[アット]tohoku.ac.jp
App Storeでのreviewをお願いします。
作者は現在、新型コロナウィルスを2分程度で検出可能な迅速検出法の構築を目指しています。これまでに新型コロナウィルスに結合するDNAアプタマーを取得し、これを複数装荷した分子を作製しているところです。このような分子はコロナルウィルスが存在すると凝集体を形成させ、これを容易に検出できるものと期待しています。これまで複数の研究助成などに応募してきましたが、残念ながら研究費の獲得に至っておりません。この研究を続けるため、私の研究室へのご寄附をご検討いただければ幸いです。
寄附金は、新型コロナウィルス関連の研究ならびにソフトウエア開発などの研究教育を目的とした活動に有効に利用させていただきます(大学によって通常の研究費と同等に管理されます)。寄附金についての本学での取り扱い、税制上の優遇措置などについてはこちらをご覧ください。
ご寄附頂ける場合は、専用の様式にご記入いただき、東北大学生命科学研究科会計係までメールにてお送りください。ご検討のほど、よろしくお願いいたします。
様式はこちらからダウンロードしてください。メールアドレスはlif-kaik[at]grp.tohoku.ac.jpです。
 大学改革の方針転換を。「REUP提案」をお読みください。
大学改革の方針転換を。「REUP提案」をお読みください。
 入試や就活で「推し研」を問いませんか?
入試や就活で「推し研」を問いませんか?
 本書では、科学的素養を7項目で整理・明文化しています。教育の質、学びの質を高め、研究力向上を。
本書では、科学的素養を7項目で整理・明文化しています。教育の質、学びの質を高め、研究力向上を。
 本書では、研究力低迷問題を分析し、解決策を提案しています。研究、教育に携わる多くの方にお読みいただけますと幸いです。解説資料平易版 ・提案資料
本書では、研究力低迷問題を分析し、解決策を提案しています。研究、教育に携わる多くの方にお読みいただけますと幸いです。解説資料平易版 ・提案資料
 QRコードをスマホで読み取って出席登録。出席管理WEBシステムです。
QRコードをスマホで読み取って出席登録。出席管理WEBシステムです。
 TraceViewer。変性ポリアクリルアミドゲル電気泳動の代わりにシーケンサーでデータを取りませんか?macOSアプリです。
TraceViewer。変性ポリアクリルアミドゲル電気泳動の代わりにシーケンサーでデータを取りませんか?macOSアプリです。
 ShortReadManager NGSデータの処理に便利なmacOSアプリです。変なアイコンですみません。
ShortReadManager NGSデータの処理に便利なmacOSアプリです。変なアイコンですみません。
 GenomeMatcher ゲノム比較機能をはじめ色々な情報処理ツールがついています。数値データをグラフィックスに変換する機能も。
GenomeMatcher ゲノム比較機能をはじめ色々な情報処理ツールがついています。数値データをグラフィックスに変換する機能も。
 GenoFinisher バクテリアゲノムをshort readだけでも決定可能です。バクテリアゲノムのフィニッシングでお困りの方はご相談ください。
GenoFinisher バクテリアゲノムをshort readだけでも決定可能です。バクテリアゲノムのフィニッシングでお困りの方はご相談ください。
 DNA配列の相補配列を計算する関数を含むエクセルシートです。
DNA配列の相補配列を計算する関数を含むエクセルシートです。
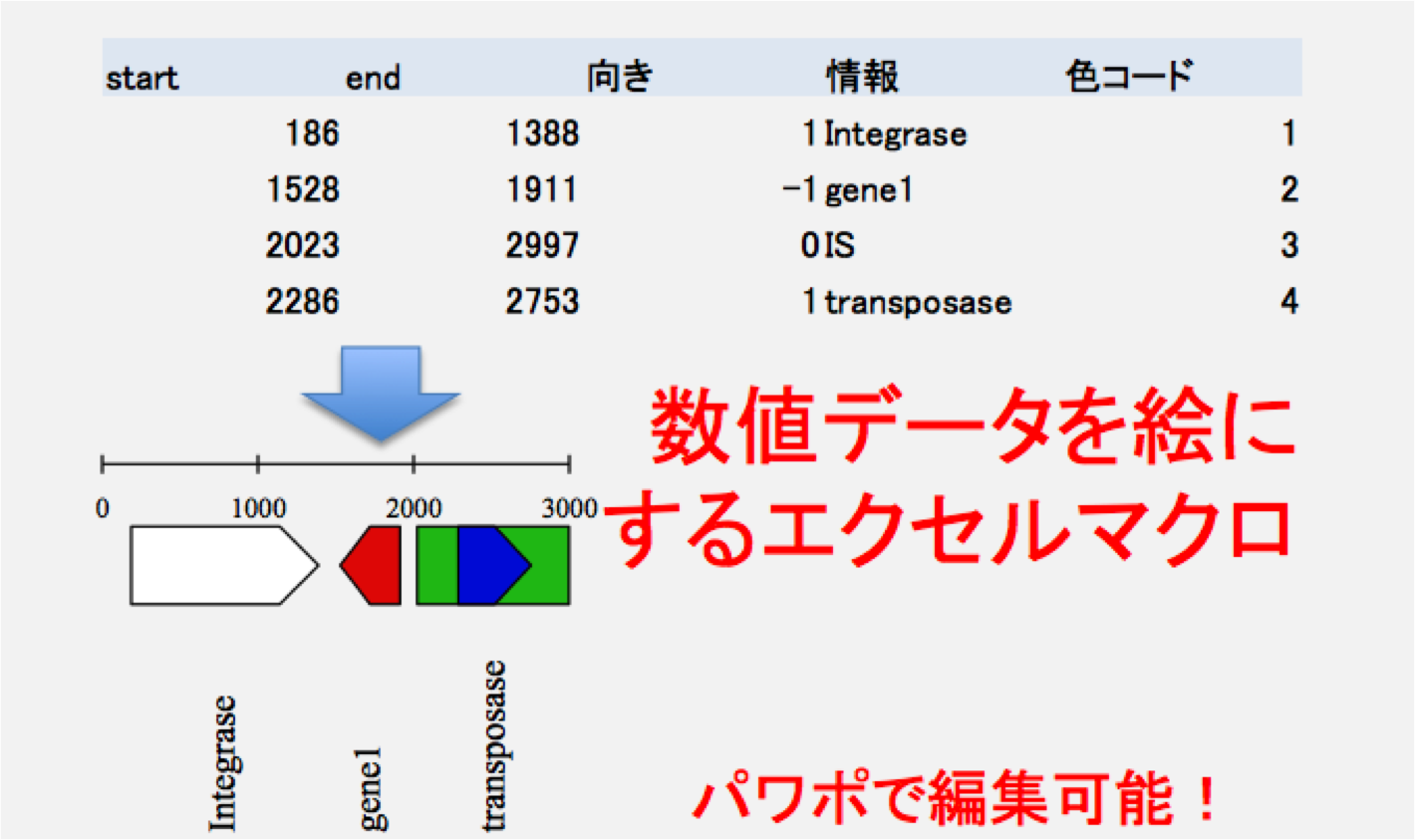 数値データを絵に変換するマクロを含むエクセルシートです。
数値データを絵に変換するマクロを含むエクセルシートです。
 数値データを元にサーキュラーマップが描けます。データ生成機能もあります。
数値データを元にサーキュラーマップが描けます。データ生成機能もあります。
 大学等研究機関での実験機器類の共用を促進するためのウエブシステムです。
大学等研究機関での実験機器類の共用を促進するためのウエブシステムです。
 DNA配列/アミノ酸配列を2次元パネルの上で動かせるソフトウエアです。配列比較も。
DNA配列/アミノ酸配列を2次元パネルの上で動かせるソフトウエアです。配列比較も。
 例の処理を簡単に済ませるあのツールです。
例の処理を簡単に済ませるあのツールです。
 iPhoneアプリです。勤務地への入域と出域時刻のログを自動的にとります。App Storeから入手できます。
iPhoneアプリです。勤務地への入域と出域時刻のログを自動的にとります。App Storeから入手できます。
 文字列集合を取り扱える便利ツールです。
文字列集合を取り扱える便利ツールです。
 GenBankファイルのデータを、エクセルシートで取り扱えるように変換するツールです。
GenBankファイルのデータを、エクセルシートで取り扱えるように変換するツールです。
 文字列を取り扱える便利ツールです。
文字列を取り扱える便利ツールです。
 作者研究室ホームページ。共同研究の提案と研究室への寄付を歓迎します。
作者研究室ホームページ。共同研究の提案と研究室への寄付を歓迎します。
作者プロフィール: 環境細菌の研究を進める一方で、様々なソフトウエアを作成、公開している。