文部科学省科学研究費補助金「新学術領域研究」
新学術領域|ゲノム・遺伝子相関
月別アーカイブ
計画研究班別アーカイブ
公募研究班別アーカイブ
旧公募研究班別アーカイブ
「研究経過報告」内を検索
「MutMap+法」 と 「MutMap-Gap法」を確立して、それぞれPLoS ONE と New Phytologistに掲載されました。
これまでに、当研究グループでは、次世代シーケンサーを用いて、迅速に突然変異体の原因遺伝子を同定する技術としてMutMap法 (Abe et al. Nature Biotechnol, 2012) を確立しました。しかし、MutMap法を直接適用できない場面があったので、MutMap法を基礎として新たに「MutMap+法」と「MutMap-Gap法」を開発し報告しました。
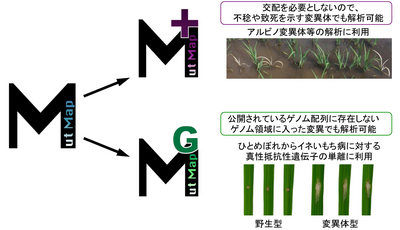
MutMap+法は、突然変異体の自殖後代において、野生型と変異体型のバルクシーケンスの結果を比較して変異体の原因遺伝子を同定する手法です。致死や不稔といった形質の解析は、交配を必要とする従来のMutMap法では困難でしたが、MutMap+法により容易になりました。さらに、MutMap+法は人工的な交配が困難な植物種にも適用できます。
MutMap-Gap法は、MutMap法とde
novo assemblyという技術を併用することで、基準ゲノム配列
(イネでは「日本晴」の配列)
に存在しないゲノム領域において変異遺伝子の同定を可能にする技術です。MutMap-Gap法を用いることで、北東北の主力イネ品種「ひとめぼれ」からいもち病に対する抵抗性遺伝子Piiを単離することに成功しました。
現在、様々な生物種において、全ゲノム配列解読が進んでいます。ゲノム解読が完了した生物種において、MutMapシリーズを活用することにより、遺伝解析が容易かつ迅速化することが期待されます。
Fekih
R, Takagi H, Tamiru M, Abe A, Natsume S, Yaegashi H, Sharma S, Sharma S,
Kanzaki H, Matsumura H, Saitoh H, Mitsuoka C, Utsushi H, Uemura A, Kanzaki E,
Kosugi S, Yoshida K, Cano L, Kamoun S, Terauchi R. (2013) MutMap+: Genetic
Mapping and Mutant Identification without Crossing in Rice. PLoS ONE 8(7):
e68529. doi:10.1371/journal.pone.0068529
Takagi
H, Uemura A, Yaegashi H, Tamiru M, Abe A, Mitsuoka C, Utsushi H, Natsume S,
Kanzaki H, Matsumura H, Saitoh H, Yoshida K, Cano LM, Kamoun S, Terauchi R.
(2013) MutMap-Gap: whole-genome resequencing of mutant F2 progeny bulk combined
with de novo assembly of gap regions identifies the rice blast resistance gene
Pii. New Phytol. doi: 10.1111/nph.12369.
論文紹介 (寺内@岩手生工研)







