文部科学省科学研究費補助金「新学術領域研究」
新学術領域|ゲノム・遺伝子相関
月別アーカイブ
計画研究班別アーカイブ
公募研究班別アーカイブ
旧公募研究班別アーカイブ
「研究経過報告」内を検索
2013年7月の記事を表示しています
6月2日から4日にかけて、班会議に参加させて頂きました。皆様の先進的なご研究内容に引き込まれ、あっという間の3日間でした。
ゲノム・遺伝子相関と銘打っている通り、ゲノムやトランスクリプトームなどのオミックス情報を対象としたご研究が多く、扱うデータの大規模化を目の当たりにしました。私はバイオインフォマティクスを専門としておりますが、バイオインフォマティクスの必要性・重要性を実感すると共に、生物学的意義のある情報をどのようにマイニングするか再考する良い機会となりました。懇親会では、皆様快く会話に加えて下さり、共同研究の話も持ち上がるなどして、まだこの世界で経験の浅い私にとって、とても貴重な経験となりました。
この度の班会議に参加する機会を頂けた事に感謝すると共に、再び班会議に参加する機会がありましたら、その際は皆様にご指導ご鞭撻の程頂ければ幸いです。
明治大学農学部生命科学科バイオインフォマティクス研究室
小林 正明 (ポストドクター)
7月25日-26日の二日間にわたり、第21回高校生のための夏休み特別講習会が東京工業大学すずかけ台キャンパスで行われました。本講習会は、生命科学の実験を通じて、この分野の研究の面白さを発見してもらうことを目的として行いました。本年度は、田中班員が「卵の中をのぞいてみよう!ーいのちがうまれるきせき」といったテーマで講師をつとめ、全国から集まった40名以上の高校生と発生学実験を行いました。
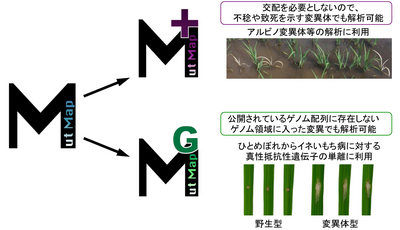
これまでに、当研究グループでは、次世代シーケンサーを用いて、迅速に突然変異体の原因遺伝子を同定する技術としてMutMap法 (Abe et al. Nature Biotechnol, 2012) を確立しました。しかし、MutMap法を直接適用できない場面があったので、MutMap法を基礎として新たに「MutMap+法」と「MutMap-Gap法」を開発し報告しました。
MutMap+法は、突然変異体の自殖後代において、野生型と変異体型のバルクシーケンスの結果を比較して変異体の原因遺伝子を同定する手法です。致死や不稔といった形質の解析は、交配を必要とする従来のMutMap法では困難でしたが、MutMap+法により容易になりました。さらに、MutMap+法は人工的な交配が困難な植物種にも適用できます。
MutMap-Gap法は、MutMap法とde
novo assemblyという技術を併用することで、基準ゲノム配列
(イネでは「日本晴」の配列)
に存在しないゲノム領域において変異遺伝子の同定を可能にする技術です。MutMap-Gap法を用いることで、北東北の主力イネ品種「ひとめぼれ」からいもち病に対する抵抗性遺伝子Piiを単離することに成功しました。
現在、様々な生物種において、全ゲノム配列解読が進んでいます。ゲノム解読が完了した生物種において、MutMapシリーズを活用することにより、遺伝解析が容易かつ迅速化することが期待されます。
Fekih
R, Takagi H, Tamiru M, Abe A, Natsume S, Yaegashi H, Sharma S, Sharma S,
Kanzaki H, Matsumura H, Saitoh H, Mitsuoka C, Utsushi H, Uemura A, Kanzaki E,
Kosugi S, Yoshida K, Cano L, Kamoun S, Terauchi R. (2013) MutMap+: Genetic
Mapping and Mutant Identification without Crossing in Rice. PLoS ONE 8(7):
e68529. doi:10.1371/journal.pone.0068529
Takagi
H, Uemura A, Yaegashi H, Tamiru M, Abe A, Mitsuoka C, Utsushi H, Natsume S,
Kanzaki H, Matsumura H, Saitoh H, Yoshida K, Cano LM, Kamoun S, Terauchi R.
(2013) MutMap-Gap: whole-genome resequencing of mutant F2 progeny bulk combined
with de novo assembly of gap regions identifies the rice blast resistance gene
Pii. New Phytol. doi: 10.1111/nph.12369.
論文紹介 (寺内@岩手生工研)
アフリカツメガエル (Xenopus laevis) は、祖先型二倍体種の異種間雑種のゲノム倍数化によって生じた異質四倍体種であることが知られています。本研究では、FISH法を用いてアフリカツメガエルの機能遺伝子染色体地図を作製し、二倍体種のネッタイツメガエル (X. tropicalis) の染色体地図と比較することによって同祖染色体対を同定し、異種間雑種の四倍体化後に生じたゲノム・染色体再編成のパターンとそのプロセスを明らかにすることを試みました。cDNAを用いた比較染色体マッピングの結果、同祖染色体9組を全て同定することができ、それぞれの遺伝連鎖群を比較した結果、2組の同祖染色体間に逆位が存在することを除けば、2種間ならびに同祖染色体間での相互転座は検出されず、異質四倍数体化後約4.000~5.000万年経過しても遺伝連鎖群は高度に保存されていることを明らかにしました。また、四倍体化後の遺伝子の欠失は非常に少なく(17%)、遺伝子レベルでの二倍体化はほとんど進んでいないことを見出しました。これらの結果は、2013年7月3日付でHeredityにオンライン publishされました。
7月6日午前、雲雀丘学園(兵庫県宝塚市)の1day collegeで、
『共生科学入門:生物共生の分子生物学と地球環境』
と題する出前向後を、高校生対象に実施してきました。
- 1
- 2









