文部科学省科学研究費補助金「新学術領域研究」
新学術領域|ゲノム・遺伝子相関
月別アーカイブ
計画研究班別アーカイブ
公募研究班別アーカイブ
旧公募研究班別アーカイブ
「研究経過報告」内を検索
【東京大】小林班の記事を表示しています
日本遺伝学会第86回大会で、小林が世話人となって、ワークショップ「
DNA の破壊と修復と再編:塩基切り出しの役割をめぐる新展開 Recent excitements in DNA base
excision」を開催しました。小林、木下哲教授(本領域、横浜市立大学)、村松正道教授(金沢大)、中別府雄作教授(九大)が講演しました。
DNA の塩基の切り出しは、DNA
塩基への損傷からの修復という観点から研究される事が多かったのですが、エピジェネティックなメチル化塩基の排除に関与することが、動物植物で明らかにな
りました(木下)。さらに、原核生物では、エピジェネティックな塩基メチル化の有無によって非自己DNA
を破壊する制限修飾系に、塩基切り出しによるものが発見されました(小林)。また、塩基切り出し酵素は、抗ウイルス活性や抗体遺伝子の多様性増幅機構への
関与が示さ
れ(村松)、さらには細胞死による発がん抑制や神経変性をもたらす事も明らかになってきました(中別府)。
DNA
の塩基の切り出しが、遺伝過程、エピジェネティックス過程、個体恒常性維持で果たす予想外に大きな役割とその分子機構について、活発なディスカッション
が、ワークショップと、その前後のナイトセミナー、懇親会でも行われました。これまでエピジェネティックス、DNA複製・修復・組換え、細胞死の
研究として進んできたものが、脱塩基サイトを巡って合流することが感じられました。
2014年8月21(木)‐24日(日)に大阪府高槻市高槻現代劇場で予定されております、日本進化学会 第16回大会においてシンポジウム「Epigenomes in evolutionエピゲノムが進化する」を開催します。皆様のご参加をお待ちします。
趣旨:
The elementary units of heredity and evolution might be epigenomes in addition to genome sequences. The innovation in genomics and OMICS now made possible to test this hypothesis. We will introduce the cutting-edge technology and results in microorganisms, plants and animals.
遺伝と進化を担うのは、「ゲノム配列」とともに、その上に重ねて書かれた「エピゲノム情報」であることを示唆する現象が蓄積している。この仮説の検証が、ゲノムとOMICS研究のイノベーションによって、今や可能になった。微生物・植物・動物での、最先端の方法と成果を紹介し、「エピゲノム工学・エピゲノム育種」への応用を展望する。
詳細はhttps://sites.google.com/site/shinka2014osaka/program/symposia/s3 をご覧ください。
公開セミナーのご案内 集団ゲノム科学 Population genomics の最先端を切り開いているDaniel Falush博士に、
以下のようにヒト集団の混合の歴史について公開セミナーをして頂きます
(Hellenthal et al. A genetic atlas of human admixture history. Science, 2014)。
皆様にも、是非ご来聴いただきたく、ご案内申し上げます。
お近くの方々にもご連絡頂ければ幸いです。
博士は、5月初めから7月末まで、東京大学特任教授として在職します。 日時:平成26年 5月23日(金)15:00 - 16:00 15:00 - 16:00, May 23rd (Fri), 2014 場所:東京大学 柏キャンパス 生命棟会議室 University of Tokyo Kashiwa Campus (Meeting Room, Biosciences Building) 東京大学 医科学研究所(地下鉄南北線、白金台駅)、2号館2階 小講義室 【遠隔配信】 Institute of Medical Science, University of Tokyo (Smaller Lecture Room, Second Floor, Building 2) (by Shirokanedai Station, Tokyo Metro) 【Remote lecture】 アクセス:http://www.ims.u-tokyo.ac.jp/imsut/jp/access/access/ キャンパスマップ:http://www.ims.u-tokyo.ac.jp/imsut/jp/access/campus/ 講師: ダニエル・ファルシュ Daniel Falush, Ph.D. マックス・プランク進化人類学研究所・シニアサイエンティスト、 東京大学大学院・新領域創成科学研究科・外国人特任教授 Senior Scientist, Max Planck Institute for Evolutionary Anthropology, Leipzig / Project Professor, GSFS, UT 演題: Inference of population structure and migration history by chromosome painting. ヒト集団構造と移動史の染色体ペインティングによる推定 概要: I will describe how fine information about ancestry can be obtained
using the chromopainter algorithm and
how this can be used to elucidate details of population structure and migration events,
using fineSTRUCTURE and globetrotter.
Examples will be presented from our recent analysis of human admixture history worldwide
and from the Peopling of the British Isles project. 集団の祖先についての詳細な情報を、コンピューター内染色体ペインティングという
アルゴリズムによって得る事ができる。それをもとに、集団構造と移動史の詳細を、
fineSTRUCTUREとglobetrotterというツールによって、明らかにできる。
世界的なヒト集団間の混合の歴史と、イギリス諸島へのヒトの定着から例をあげる。 http://www.ncbi.nlm.nih.gov/pubmed/24531965 世話人 : 鈴木穣、新領域創成科学研究科・情報生命科学専攻・教授 E-mail : ysuzuki@k.u-tokyo.ac.jp Tel: 04-7136-3607 小林一三、新領域創成科学研究科・メディカルゲノム専攻・教授 E-mail : ikobaya@ims.u-tokyo.ac.jp Tel: 03-5449-5326
集団ゲノム科学 Population genomics の最先端を切り開いているDaniel Falush博士に、以下のように細菌ゲノムについて公開セミナーをして頂きます。皆様にも、是非ご来聴いただきたく、ご案内申し上げます。お近くの方々にもご連絡頂ければ幸いです。博士は、5月初めから7月末まで、東京大学特任教授として在職します。
日時:平成 26 年5月2 日(金)16:30 - 18:00 16:30 - 18:00, May 2nd (Fri), 2014 場所:東京大学柏キャンパス 生命棟セミナー室3【遠隔配信】 University of Tokyo Kashiwa Campus (Seminar Room 3, Biosciences Building)【Remote lecture】 東京大学 医科学研究所(地下鉄南北線、白金台駅)、2号館2階小講義室 Institute of Medical Science, University of Tokyo (Smaller Lecture Room, Second Floor, Building 2) (by Shirokanedai station, Tokyo Metro) アクセス:http://www.ims.u-tokyo.ac.jp/imsut/jp/access/access/ キャンパスマップ:http://www.ims.u-tokyo.ac.jp/imsut/jp/access/campus/ 講師:ダニエル・ファルシュ Daniel Falush, Ph.D. マックス・プランク進化人類学研究所・シニアサイエンティスト、新領域創成 科学研究科・外国人特任教授 Senior Scientist, Max Planck Institute of Evolutionary Anthropology / Project Professor, GSFS 演題:Evolutionary genomics of rapidly recombining bacteria. 高速組換え細菌の進化ゲノム学 概要: I will describe recent work on how campylobacter has evolved in response to the challenges of the agricultural environment. I will then describe the very different setting of ocean bacteria Vibrio parahaemolyticus that live in the ocean but also colonize animal hosts. 農業環境の変化に応じて、カンピロバクターがどう進化してきたかについて、最近の 成果を紹介する。次に、大洋に生存しながら動物にも住み着ける海洋細菌である腸炎 ビブリオの全く異なる状況について述べる。
世話人:小林一三 (75326, ikobaya@ims.u-tokyo.ac.jp)
タンパク質の合成(翻訳)は、メッセンジャーRNA(mRNA)のリボソームによる認識によっておこります。原核生物では、mRNAの5'の非翻訳領域にあるシグナル配列(SD配列)が、リボソームの小サブユニットのRNAの3'端(アンチSD配列、コアモチーフ3'CCUCC) と相補的な塩基対を作る、SD(Shine-Dalgarno)相互作用が起きます。
私たちは、藍藻(シアノバクテリア)由来の内部共生体が葉緑体などの色素体に進化する過程で、この相互作用がどうなったかを調べました。SD相互作用の消失が、緑藻 、ユーグレナ藻、アピコンプレックス門の色素体で、並行して起きていました。それらの色素体ゲノムの著しい縮小と関係するのでしょう。
古典的な SD 相互作用 (3′CCUCC/5′GGAGG (rRNA/mRNA)) が、 変則的な SD 相互作用 (3′CCCU/5′GGGA あるいは 3′CUUCC/5′GAAGG) にとりかわっている場合が、緑藻とユーグレナ藻で、発見されました。それは、rRNA 側のモチーフ配列と mRNA側のシグナル配列の双方の、足並みを揃えた変化によって起きていました(図:ユーグレナ藻)。
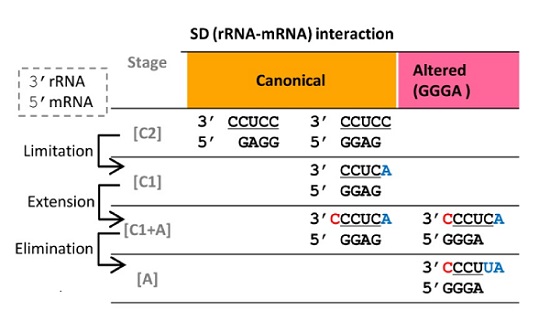
rRNAとmRNAのこのような共進化は、 遺伝情報発現のしくみの予想外な進化的可塑性を示しています。
Kyungtaek Lim, Ichizo Kobayashi, and Kenta Nakai. Alterations in rRNA-mRNA interaction during plastid evolution. Molecular Biology and Evolution. (2014) doi: 10.1093/molbev/msu120 [Journal]







