文部科学省科学研究費補助金「新学術領域研究」
新学術領域|ゲノム・遺伝子相関
月別アーカイブ
計画研究班別アーカイブ
公募研究班別アーカイブ
旧公募研究班別アーカイブ
「研究経過報告」内を検索
【岩手生工研】寺内班の記事を表示しています
植物病原体は、宿主植物への感染時にエフェクターと呼ばれるタンパク質を分泌する。エフェクターは様々な戦略で宿主植物の防御反応を抑制したり、細胞環境を変化させたりして病原体の感染を成立させる役割を担っている。近年、植物病原菌のゲノム配列解読により、様々な病原菌のエフェクタ−の同定が可能となっている。新奇のエフェクタ−タンパク質の同定およびその機能解析は、病原菌の病原性や宿主植物の防御機構を理解する上で重要である。
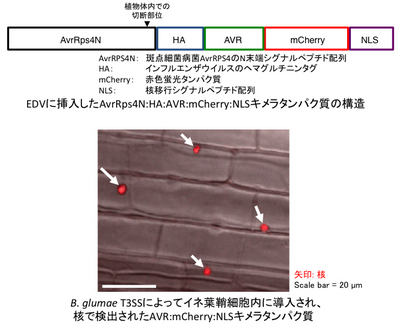
Sohnらは、Effector Detector Vector(EDV)と斑点細菌病菌のIII型分泌機構(T3SS)を利用し、双子葉植物におけるべと病菌エフェクタ−の探索および機能解析に成功した(Sohn et al. 2007 Plant Cell 19: 4077-4090)。本研究では、イネもみ枯細菌病菌(Burkholderia glumae)T3SSを利用し、イネいもち病菌エフェクタ−AVR-Pik、AVR-Piiの単子葉植物細胞内への導入および機能解析を行った。
蛍光タンパク質と核移行シグナルを融合させたAVRをEDVにクローン化してB. glumae を形質転換し、イネ、コムギ、オオムギおよびNicotiana benthamianaに接種した結果、接種組織細胞内の核において蛍光シグナルが検出された。また、B. glumae T3SSによって導入されたAVR-PikとAVR-Piiは、対応する抵抗性遺伝子を保有するイネ品種において非病原性を示した。B. glumaeを非宿主植物N. benthamianaに接種すると過敏感細胞死(HR)を生じるが、導入されたAVR-PikはそのHRを遅延させた。本研究により、単子葉植物におけるエフェクターの探索および機能解析の目的でB. glumae T3SSを利用できる可能性が示された。
Sharma, S., Sharma, S., Hirabuchi, Yoshida, K., Fujisaki, K., Ito, A., Uemura, A., Terauchi, R., Kamoun, S., Sohn, K.H., Jones, J.D.G. and Saitoh, H.
The Plant Journal (2013) 74: 701-712
http://onlinelibrary.wiley.com/doi/10.1111/tpj.12148/full
論文紹介 (齋藤@岩手生工研)
これまでにDNAマーカーを用いた連鎖解析により,イネいもち病に対する抵抗性遺伝子座が同定されてきました。しかし、従来の連鎖解析を用いたQTL同定方法では、交配に用いた両品種間で多型を示すDNAマーカーを作成するステップにおいて多くの労力および時間を必要としました。今回、論文に掲載されたQTL-seq法は、次世代シーケンサーを用いることで、DNAマーカーを作成することなく迅速にQTL同定を可能にする技術です。
本方法を用いることで、従来のDNAマーカーを用いた連鎖解析によるQTL同定法と比較して、迅速かつ低コストでQTLを同定することが可能になりました。
 QTL-seq: Rapid mapping of quantitative
trait loci in rice by whole genome resequencing of DNA from two bulked
populations
QTL-seq: Rapid mapping of quantitative
trait loci in rice by whole genome resequencing of DNA from two bulked
populations
Hiroki Takagi, Akira Abe, Kentaro Yoshida, Shunichi Kosugi, Satoshi Natsume, Chikako Mitsuoka, Aiko Uemura, Hiroe Utsushi, Muluneh Tamiru, Shohei Takuno, Hideki Innan, Liliana M. Cano, Sophien Kamoun and Ryohei Terauchi
The Plant Journal, 2013, DOI: 10.1111/tpj.12105
論文紹介 (寺内@岩手生工研)
生物間相互作用は個々の生物にどのような影響を与え共進化をもたらしているのでしょうか? 病原菌に対する宿主植物の防御メカニズムは、宿主植物と病原菌との相互作用によって変化します。私たちは、いもち病菌から分泌されてイネの細胞に送り込まれる非病原力遺伝子(AVR)タンパク質と、対応するイネの抵抗性遺伝子(R)タンパク質とが、直接的タンパク質相互作用し、アミノ酸変異による両者の結合度の違いが抵抗性の程度を決定していることを明らかにしました。その研究成果はThe Plant Journalに掲載されました。
抵抗性タンパク質Pikは2種のCC-NBS-LRRであるPik1とPik2から構成されます(Ashikawa et al.2008,Genetics 180:2267)が、その一方のPik1が、直接的タンパク質相互作用によっていもち病菌AVR-Pikを認識することが、本研究によって明らかとなりました。さらに、AVR-Pikと結合するPik1のタンパク質領域は、アリル間で変異に富む領域であることも分かりました。また、いもち病菌由来のAVR-Pik、及びイネの抵抗性遺伝子Pikには、それぞれ多数のアリルがあり、アミノ酸変異をもたらすDNA変異で異なっています。このAVR-PikとPikのアリル間での直接的タンパク質相互作用の結合度の違いが、アリル間の特異的抵抗性認識に影響をもたらすことも明らかとなりました。以上の結果から、イネ抵抗性遺伝子Pikは、いもち病菌AVR-Pik産物とのタンパク質直接的相互作用を介して抵抗性を獲得し、AVR-Pikと共進化していること、Pik1の変異がAVR-Pikアリルに対する抵抗性認識に重要な役割を担っていることが明らかになりました。
Arms
race co-evolution of Magnaporthe
oryzae AVR-Pik and rice Pik genes
driven by their physical interactions
H. Kanzaki,
K. Yoshida, H. Saitoh, K. Fujisaki, A. Hirabuchi, L. Allaux, E. Fournier, D.
Tharreau, R. Terauchi
The Plant
Journal, 2012 DOI:1365-313X.2012.05110.x.
http://www.ncbi.nlm.nih.gov/pubmed?term=22805093
論文紹介 (寺内@岩手生工研)
植物病原体は、宿主植物への感染時にエフェクターと呼ばれるタンパク質を分泌する。エフェクターは様々な戦略で宿主植物の防御反応を抑制し、病原体の感染を成立させる役割を担っている。
いもち病はイネの最重要病害であるため、イネのいもち病抵抗性機構およびいもち病菌のイネへの感染機構の解明は非常に重要な課題である。しかしながら、いもち病を含む糸状菌病において、エフェクター関連研究の成果は、未だ乏しい状況である。
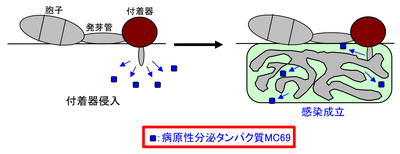
そこで、イネいもち病菌由来エフェクターを探索するために、感染初期に発現している78個の推定分泌タンパク質遺伝子の大規模破壊解析を行った。大多数の遺伝子破壊はいもち病菌の生長、胞子形成および病原性に影響を及ぼさなかった。一つだけ例外があり、その遺伝子をMC69と名付けた。mc69変異体はイネとオオムギへの病原性が著しく低下していた。mc69変異体は培地上での生長、胞子形成および宿主植物上での付着器(病原菌が植物に侵入する際に形成する器官)形成率において野生型と比較して差異を示さなかった。また、MC69タンパク質は実際にいもち病菌から分泌されることが明らかとなり、これらの結果はMC69が宿主植物との相互作用において重要な役割を果たすことを示唆している。さらに、ウリ類炭疽病菌におけるMC69相同遺伝子の欠失は、キュウリとNicotiana benthamianaへの病原性を低下させた。以上の結果より、MC69は、単子葉および双子葉植物にそれぞれ病原性を示すイネいもち病菌およびウリ類炭疽病菌の感染に共通して必要な病原性分泌タンパク質であることが明らかとなった。
 Saitoh, H., Fujisawa,
S., Mitsuoka, C., Ito, A., Hirabuchi, A., Ikeda, K., Irieda, H., Yoshino, K.,
Yoshida, K., Matsumura, H., Tosa, Y., Win, J., Kamoun, S., Takano, Y., and
Terauchi, R.
Saitoh, H., Fujisawa,
S., Mitsuoka, C., Ito, A., Hirabuchi, A., Ikeda, K., Irieda, H., Yoshino, K.,
Yoshida, K., Matsumura, H., Tosa, Y., Win, J., Kamoun, S., Takano, Y., and
Terauchi, R.
PLoS Pathogens (2012) 8: e1002711
http://www.plospathogens.org/article/info:doi/10.1371/journal.ppat.1002711
論文紹介 (齋藤@岩手生工研)
次世代シーケンサーの登場により、研究手法が大きく様変わりしていることはご存じの通りと思います。今回、本新学術領域研究「寺内班」により、さらに画期的な次世代シーケンサーを使った手法がNature Biotechnologyに発表されましたので、その内容を簡単に紹介します。
従来法では、突然変異体の原因遺伝子の同定には、SNPsの存在する系統と掛け合わせて、F2世代の集団からバルクでDNAを回収し、次世代シーケンサーを用いてSNPsのタイプを読み取り、注目する表現型との連鎖解析をするものでした。今回、寺内班により改良されたMutMap法では、SNPsにはEMS処理で置換された塩基を使います。従って、交配する場合はEMS処理した同一系統の野生型を使用します。
これにより、扱うSNPsの数が圧倒的に減り、解析が容易になりました。また、F2集団は少なくてすむため、広大な栽培スペースが必要であった作物にはうってつけです。同一系統内で解析するため、従来法のような表現型のバックグラウンドノイズが少なく、これまで困難であった小さな表現型の差異も容易に検出できます。また、原理的には、交配によらず解析することが可能であるため、交配が困難な生物でもM2世代が得られるなら解析が可能です。
今後、突然変異体の原因遺伝子の同定には、MutMap法が使われるようになり、従来法は過去のものとなる革命的な進歩と思われます。 Genome sequencing reveals agronomically important loci in rice using MutMap.
Genome sequencing reveals agronomically important loci in rice using MutMap.
Abe, A., Kosugi, S., Yoshida, K., Natsume, S., Takagi, H., Kanzaki, H., Matsumura, H., Yoshida, K., Mitsuoka, C., Tamiru, M., Innan, H., Cano, L., Kamoun, S., and Terauchi, R.
Nature Biotechnol (2012) 30: 174-178.
http://www.nature.com/nbt/journal/v30/n2/abs/nbt.2095_ja.html
論文紹介(木下@奈良先端大)







